♦ 带有致病性的肠道细菌可以诱导人类肠道细胞发生突变

图源:论文截图
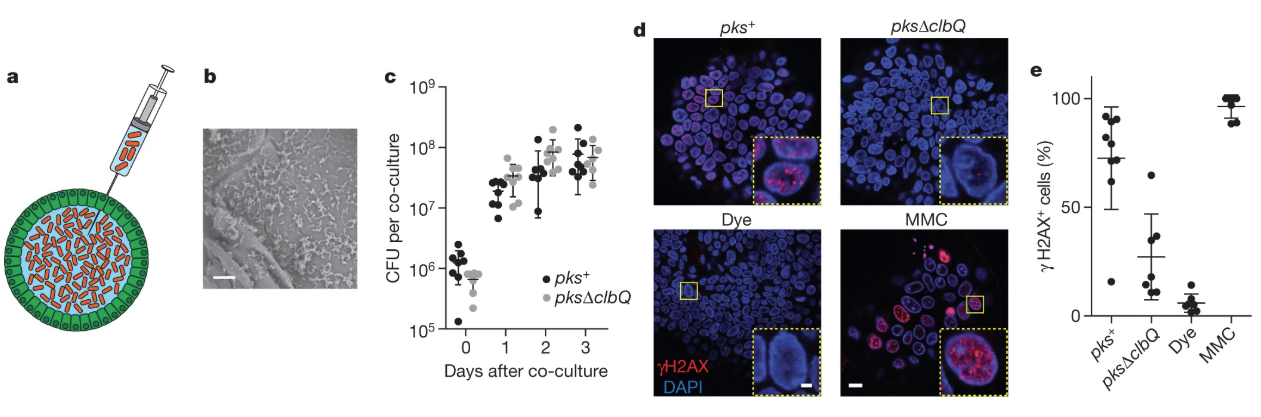
图源:文章Figure
尽管多种肠道微生物与结直肠癌的发展有关,但尚未证明细菌在致癌突变的发生中具有直接作用。来自荷兰KNAW研究院的Hans Clevers团队发现将pks+(编码大肠菌素酶)大肠杆菌与人类肠道类器官共培养,可以诱导类器官发生突变,且这些突变特征与两个独立队列的肠癌基因组突变特征相似。这是首次发现带有致病性的肠道细菌可以诱导人类肠道细胞发生突变的直接证据,也提示我们或许有来自其他微生物的其他蛋白或活性物质,可以直接导致人类细胞的DNA损伤。
https://www.nature.com/articles/s41586-020-2080-8
评论:孙莹莹
♦ 利用SVM模型来识别神经肽
图源:论文截图
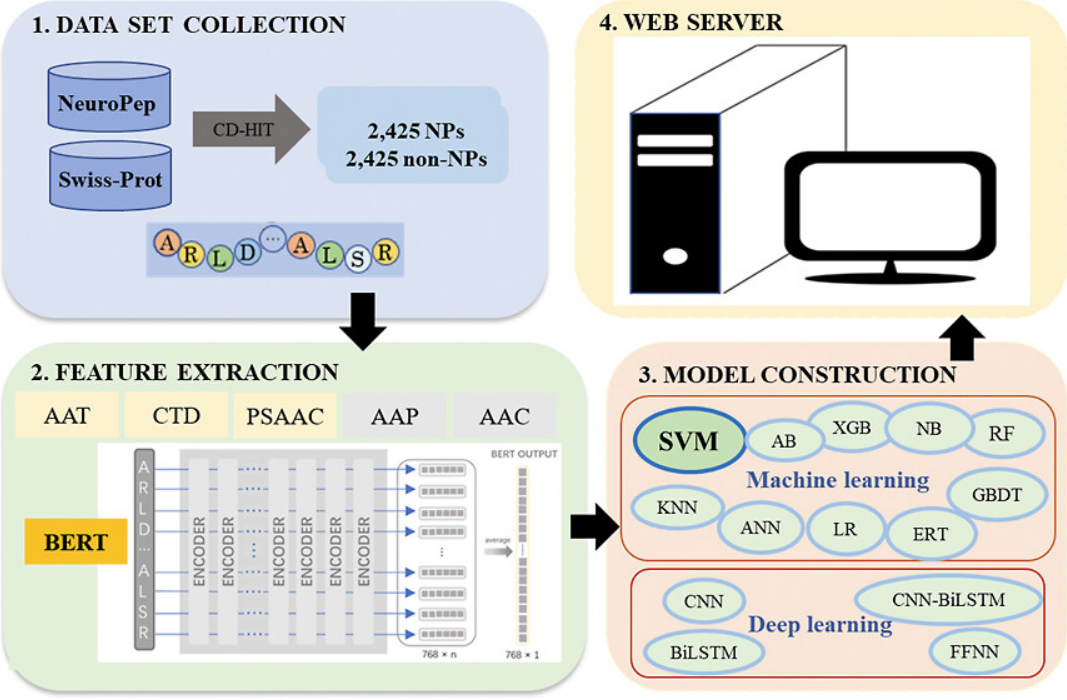
图源:文章Abstract
神经肽是多种神经元分泌的多肽,具有神经调节、神经递质或激素功能。神经肽的鉴定对于理解生物过程的机制以及药物设计有着很大的帮助。目前已经开发出一定的实验方法,可以将实验光谱与已知的神经肽数据库进行比较,识别神经肽;也可根据基质辅助激光解吸电离质谱、电喷雾质谱来发现新的神经肽。随着神经肽数据的增多,神经肽数据库逐渐完善起来,基于这些数据库出现了许多神经肽以及其性质的预测的算法。本实验中作者提出了NeuroPpred-SVM,通过向模型中输入AAC、AAP、AAT、CTD、PSAAC以及BERT嵌入特征,利用SVM模型来识别神经肽,AUROC值为0.966,优于原有算法,在跨物种预测层面也取得较好效果。
https://doi.org/10.1021/acs.jproteome.2c00363
♦ 关于肥胖的标记物的工作

图源:论文截图
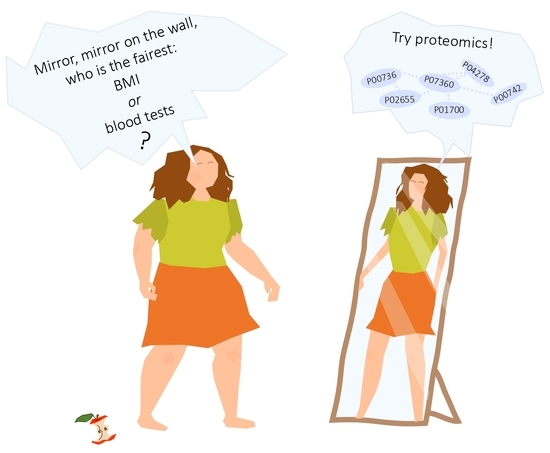
图源:文章Abstract
一篇来自俄罗斯生化所发表在journal of personalized medicine的关于肥胖的标记物的工作,收集了五类BMI分组的104人的plasma样品,先证明普通血液无肥胖预测力,然后建立了一个基于5个蛋白的LASSO回归模型预测BMI达到MAE=3.2,说明了蛋白质组应用于个体化医疗的可行性。(graphical abstract很形象)

图源:论文截图
图源:文章Figure
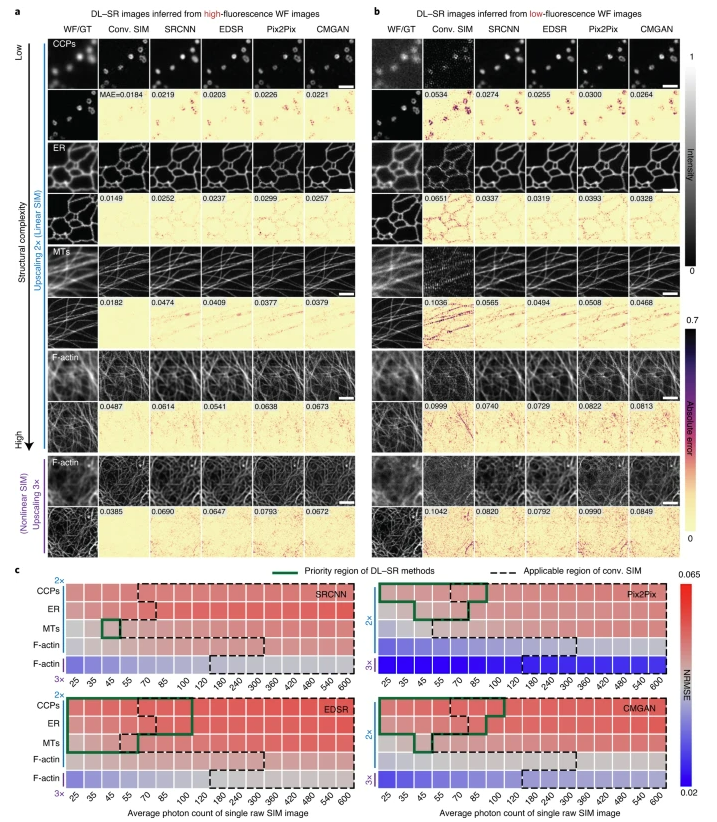
中科院生物物理研究所李栋老师团队2021年nmethods: 用低清显微镜(LowResolution)还原超高清高清显微镜(SuperResolution)图片深度学习新方法的工作。
1) 先用低清和高清显微镜产生了 50 对的低清-高清的数据集
2) 然后用这些数据评估了现有的几种低清恢复高清的模型 , 评估标准有模型结构复杂性、信噪比和放大因子
3) 展示了深度傅里叶通道注意力网络 ,它利用不同特征之间的频率内容差异来学习,对低信噪比的图像有更好的重建性
4) 最后用多色活细胞成像实验中展示了他们算法和真正的超高精度分辨率显微镜得到的图像差不多的质量,另外做了两个一个线粒体嵴和类核以及细胞器和细胞骨架的相互作用的实验应用
♦ 使用光学显微镜对生物过程进行成像
图源:论文截图
图源:文章Abstract
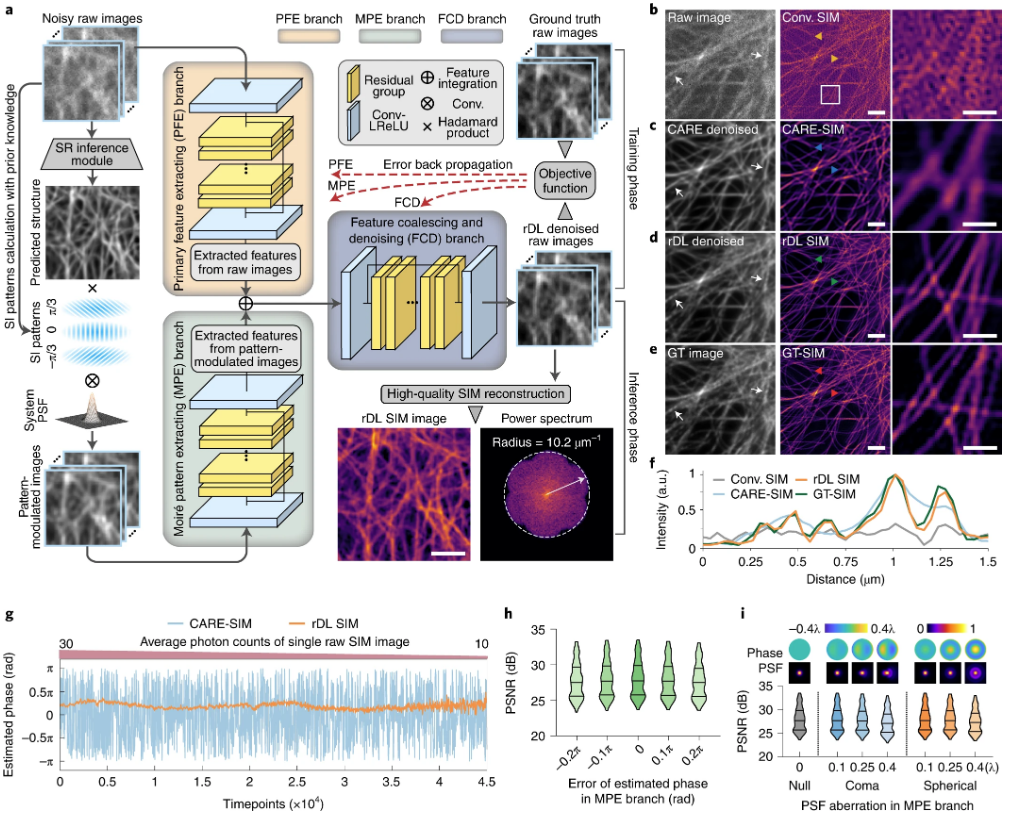
使用光学显微镜对生物过程进行成像的目标是以最少的侵入性获取最多的时空信息。深度神经网络显着改进了光学显微镜,包括图像超分辨率和恢复,但仍有很大的潜力。在这项研究中,团队通过结合照明模式的先验知识,为结构化照明显微镜和晶格光片显微镜(LLSM)开发了合理化深度学习(rDL),从而合理地引导网络对原始图像进行去噪。rDL去噪模块以原始的SIM图像以及调制图像为输入,来从输入信息中提取层次特征,再把这些特征重构为SIM图像。架构分为三个部分:PFE、MPE以及FCD,每个分支都基于残差学习以及注意力机制。团队证明了rDL方法可以将分辨率信息提升为其它计算方法的10倍以上。并通过表征运动纤毛与细胞器的互作、膜和无膜细胞器之间的联系以及活细胞4D成像来证明成像方法的可行性。
♦ 有监督的变分贝叶斯框架SPEAR

图源:论文截图
疑似Steven Gygi的儿子Jeremy Gygi发表了一篇BioRxiv,提出了一个有监督的变分贝叶斯框架SPEAR。这个框架提出的动机是,无监督因素模型由于因子构建和预测模型学习之间的分离,所得到的因子对于预测任务来说是次优的。需要一个有效利用相应的监督因素模型,同时考虑组学的结构异质性。SPEAR框架将多组学数据分解为具有预测能力的潜在因素。该方法可以自适应地确定因子秩,强调因子结构、数据相关性和特征稀疏性。SPEAR改进了合成示例中潜在因素的重建,其与Lasso、DIABLO、MOFA相比提高了新冠肺炎严重程度和乳腺癌症肿瘤亚型的预测准确性。
♦ 带有致病性的肠道细菌可以诱导人类肠道细胞发生突变

图源:论文截图

图源:文章Figure
尽管多种肠道微生物与结直肠癌的发展有关,但尚未证明细菌在致癌突变的发生中具有直接作用。来自荷兰KNAW研究院的Hans Clevers团队发现将pks+(编码大肠菌素酶)大肠杆菌与人类肠道类器官共培养,可以诱导类器官发生突变,且这些突变特征与两个独立队列的肠癌基因组突变特征相似。这是首次发现带有致病性的肠道细菌可以诱导人类肠道细胞发生突变的直接证据,也提示我们或许有来自其他微生物的其他蛋白或活性物质,可以直接导致人类细胞的DNA损伤。
https://www.nature.com/articles/s41586-020-2080-8
评论:孙莹莹
♦ 利用SVM模型来识别神经肽

图源:论文截图

图源:文章Abstract
神经肽是多种神经元分泌的多肽,具有神经调节、神经递质或激素功能。神经肽的鉴定对于理解生物过程的机制以及药物设计有着很大的帮助。目前已经开发出一定的实验方法,可以将实验光谱与已知的神经肽数据库进行比较,识别神经肽;也可根据基质辅助激光解吸电离质谱、电喷雾质谱来发现新的神经肽。随着神经肽数据的增多,神经肽数据库逐渐完善起来,基于这些数据库出现了许多神经肽以及其性质的预测的算法。本实验中作者提出了NeuroPpred-SVM,通过向模型中输入AAC、AAP、AAT、CTD、PSAAC以及BERT嵌入特征,利用SVM模型来识别神经肽,AUROC值为0.966,优于原有算法,在跨物种预测层面也取得较好效果。
https://doi.org/10.1021/acs.jproteome.2c00363
♦ 关于肥胖的标记物的工作

图源:论文截图

图源:文章Abstract
一篇来自俄罗斯生化所发表在journal of personalized medicine的关于肥胖的标记物的工作,收集了五类BMI分组的104人的plasma样品,先证明普通血液无肥胖预测力,然后建立了一个基于5个蛋白的LASSO回归模型预测BMI达到MAE=3.2,说明了蛋白质组应用于个体化医疗的可行性。(graphical abstract很形象)

图源:论文截图

图源:文章Figure
中科院生物物理研究所李栋老师团队2021年nmethods: 用低清显微镜(LowResolution)还原超高清高清显微镜(SuperResolution)图片深度学习新方法的工作。
1) 先用低清和高清显微镜产生了 50 对的低清-高清的数据集
2) 然后用这些数据评估了现有的几种低清恢复高清的模型 , 评估标准有模型结构复杂性、信噪比和放大因子
3) 展示了深度傅里叶通道注意力网络 ,它利用不同特征之间的频率内容差异来学习,对低信噪比的图像有更好的重建性
4) 最后用多色活细胞成像实验中展示了他们算法和真正的超高精度分辨率显微镜得到的图像差不多的质量,另外做了两个一个线粒体嵴和类核以及细胞器和细胞骨架的相互作用的实验应用
♦ 使用光学显微镜对生物过程进行成像

图源:论文截图

图源:文章Abstract
使用光学显微镜对生物过程进行成像的目标是以最少的侵入性获取最多的时空信息。深度神经网络显着改进了光学显微镜,包括图像超分辨率和恢复,但仍有很大的潜力。在这项研究中,团队通过结合照明模式的先验知识,为结构化照明显微镜和晶格光片显微镜(LLSM)开发了合理化深度学习(rDL),从而合理地引导网络对原始图像进行去噪。rDL去噪模块以原始的SIM图像以及调制图像为输入,来从输入信息中提取层次特征,再把这些特征重构为SIM图像。架构分为三个部分:PFE、MPE以及FCD,每个分支都基于残差学习以及注意力机制。团队证明了rDL方法可以将分辨率信息提升为其它计算方法的10倍以上。并通过表征运动纤毛与细胞器的互作、膜和无膜细胞器之间的联系以及活细胞4D成像来证明成像方法的可行性。
♦ 有监督的变分贝叶斯框架SPEAR

图源:论文截图
疑似Steven Gygi的儿子Jeremy Gygi发表了一篇BioRxiv,提出了一个有监督的变分贝叶斯框架SPEAR。这个框架提出的动机是,无监督因素模型由于因子构建和预测模型学习之间的分离,所得到的因子对于预测任务来说是次优的。需要一个有效利用相应的监督因素模型,同时考虑组学的结构异质性。SPEAR框架将多组学数据分解为具有预测能力的潜在因素。该方法可以自适应地确定因子秩,强调因子结构、数据相关性和特征稀疏性。SPEAR改进了合成示例中潜在因素的重建,其与Lasso、DIABLO、MOFA相比提高了新冠肺炎严重程度和乳腺癌症肿瘤亚型的预测准确性。






