人体内,藏着刀光剑影的微生物江湖。
肠道是人体最大的微生态系统,存在多达100万亿的微生物,参与人体免疫系统的成熟,并具有重要的免疫功能。
口腔是人体五大微生物生态系统之一,拥有数百到数千种物种和菌型。庞大的微生物群在这一隅间搅弄风云,掀起江湖波澜:口腔细菌的平衡变化不仅涉及口臭、龋齿和牙周炎等口腔疾病,还包括影响呼吸系统、循环系统、内分泌系统甚至恶性肿瘤的全身性疾病。
胃癌,是全球最常见的消化系统恶性肿瘤之一。据全球癌症观察站GLOBOCAN的统计数据,2020年,胃癌在全球新发病例中超过一百万例,估计有769,000例死亡。目前,我们迫切需要一种精确且广泛适用的方法来识别存在高胃癌风险的个体。
舌苔(tongue coating)是舌背上的可见层,包括死亡的上皮细胞、血液代谢物、微生物、鼻后区和牙龈的分泌物以及唾液。舌苔是口腔微生物的栖息地,由于口腔直接连接到消化道,因而舌苔为评估胃的状况提供了宝贵的视角。
早在两千多年前,中医便有 “舌诊”(tongue diagnosis),利用舌苔颜色和厚度变化作为个体健康状况的指标。《黄帝内经》 、《伤寒杂病论》等医学基础经典中已经概述了舌理论的许多重要方面。[1]
图1 元代诊断学著作《敖氏伤寒金镜录》中的三十六舌图
如今,“舌诊” 已经退居二线。但现代研究人员正在分子层面探究舌头所隐藏的健康和疾病的大量信息。
之前的大多数研究都是基于宏基因组学在基因组水平上描述舌苔特征。随着宏蛋白组学(metaproteomics)的发展,研究人员现在能够在蛋白质水平上分析微生物在疾病过程中的贡献。
目前,对口腔宏蛋白质组学的研究仍处于初期阶段,主要集中在牙龈龋齿或口腔恶性肿瘤上。只有2022年10月份的一项研究揭示了口腔宏蛋白质组学与肺癌之间的关系,并且没有研究关于胃癌患者的口腔宏蛋白质组学 [2]。我们对于胃癌患者舌苔发展背后的机制仍然不够了解。

图2 论文截图
2024年1月8日,中国科学院大学附属肿瘤医院(浙江省肿瘤医院)程向东、徐志远团队联合西湖大学蛋白质组大数据实验室郭天南团队,合作在Microbiome上发表了新的论文 In-depth metaproteomics analysis of tongue coating for gastric cancer: a multicenter diagnostic research study。西湖欧米参与该研究的部分数据处理工作。
文章通过采集胃癌患者和非胃癌患者的舌苔样本(从345个舌苔样本中测得1432个人源蛋白和13,780个微生物蛋白),利用宏蛋白质组学分析揭示了舌苔中人类和微生物蛋白的差异,发现了与胃癌风险相关的微生物菌群,并建立了基于舌苔微生物蛋白的机器学习模型,为胃癌的非侵入性诊断提供了新的方法,适用于大规模应用。
研究样本及方法:
研究共收集了来自中国五个独立研究中心的180名胃癌患者和185名非胃癌患者的417个舌苔样本。经过质量控制,最终纳入233个ZJC(浙江省肿瘤医院)队列和112个多中心队列的样本进行后续分析。
利用压力循环技术和数据非依赖性采集(PCT-DIA)质谱法提取和鉴定舌苔蛋白质。通过时间序列队列调查舌苔蛋白的时间稳定性。此外,使用随机梯度提升算法(stochastic gradient boosting model (GBM))构建机器学习模型,基于舌苔微生物蛋白鉴别胃癌患者。
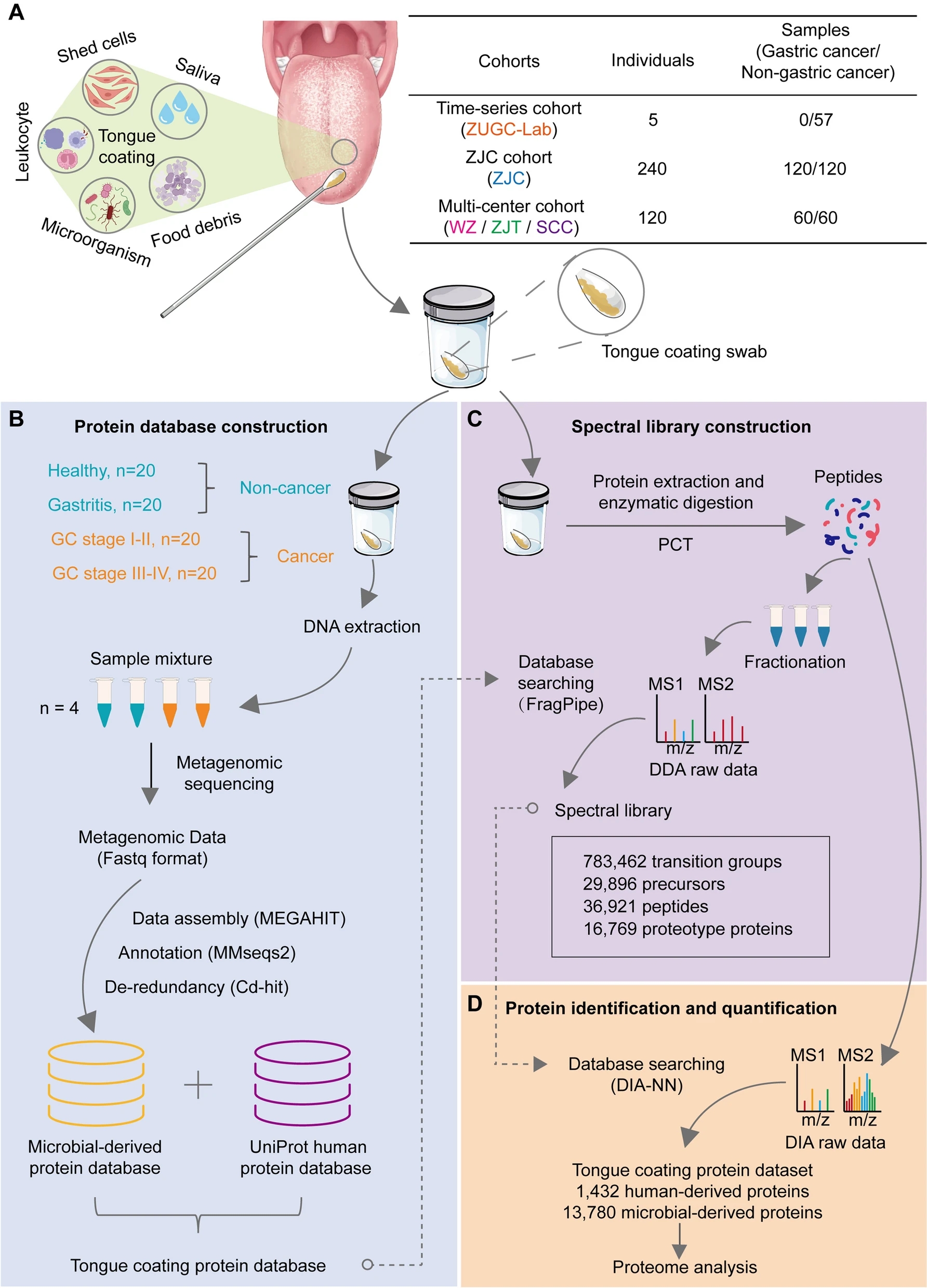
图3 研究示意图 A 舌苔的主要成分和本研究中的队列信息;B 舌苔蛋白数据库的构建过程;C 舌苔蛋白谱库的构建;D 基于PCT-DIA的舌苔蛋白鉴定和定量工作流程
结论1
舌苔蛋白质组的功能特征
通过比较ZJC队列和多中心队列的蛋白质组数据,研究人员发现舌苔中微生物蛋白的数量约为人源蛋白的九倍(1432 种人类来源蛋白质、13,780种微生物来源蛋白质)。人源蛋白主要富集在细胞应激和中性粒细胞脱颗粒等途径,而微生物蛋白则主要富集在碳代谢、氨基酸合成等途径。
结论2
舌苔微生物的分类
研究人员利用Unipept对所有舌苔样本中的微生物进行了分类,结果显示舌苔微生物主要为细菌,ZJC队列中鉴定出25个门(以杆菌门、拟杆菌门、假单胞菌门、放线菌门和梭菌门为主)。多中心队列与ZJC队列在门、纲、目、科、属和种水平上具有相似的舌苔微生物多样性。
结论3
胃癌患者舌苔微生物结构的变化
通过比较胃癌组和非癌组的样本,以及不同分期的癌症患者,发现它们在多样性上没有显著差异。通过回归分析发现,在物种水平上,某些微生物与胃癌风险相关,如 Schaalia odontolytica 菌与较高的胃癌风险相关,而 Acetatifactor 菌和 Stomatobaculum longum 菌与ZJC 队列中较低的胃癌风险相关。在多中心队列中,Neisseria brasiliensis 菌被发现是胃癌的危险因素,Jeotgalibace porci 菌则与较低的胃癌风险相关。
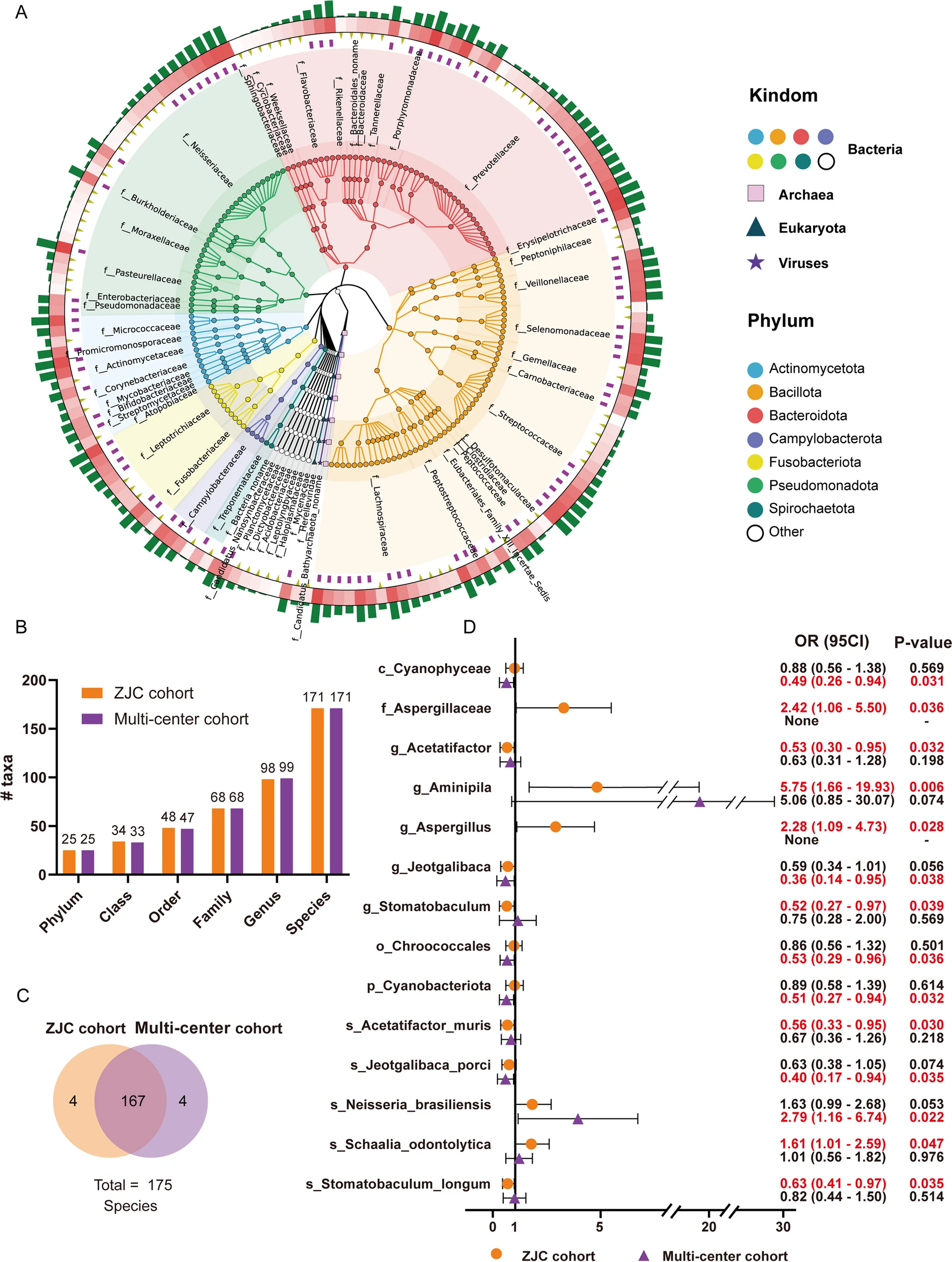
图4 胃癌患者舌苔微生物群的分类概述和结构变化 A ZJC队列样本中舌苔微生物群的分类树;B ZJC 队列和多中心队列中鉴定出的细菌数量;C ZJC 队列和多中心队列中鉴定出的细菌种类总数的比较;D ZJC 队列和多中心队列中与胃癌风险相关的细菌
结论4
胃癌患者舌苔蛋白的功能变化
为进一步研究胃癌患者舌苔蛋白的变化,研究人员对ZJC队列和多中心队列中的人类蛋白进行了差异蛋白分析。分析结果显示,ZJC 队列的胃癌患者中共有 12 种蛋白质上调,9 种蛋白质下调,两个队列的胃癌患者中共同下调的蛋白有5种(KRT2、KRT9、DCD、EWSR1 和 CACNA1G ,其中KRT2 和 KRT9 是构成舌苔的重要角蛋白)。
接下来,研究人员对胃癌患者舌苔中所有下调的人类蛋白进行了功能富集分析。结果发现这些蛋白质主要与舌上皮的生长和分化有关,这意味着胃癌患者舌表面的物理屏障被削弱。进一步的分析表明,人源蛋白与微生物蛋白之间存在相关性,且在胃癌患者中COG1136下调,这是ABC转运蛋白的一部分,提示了舌黏膜的防御能力下降。
结论5
基于微生物蛋白的胃癌筛查模型
研究人员发现,舌苔微生物蛋白的个体内稳定性和个体间变异性显示出识别胃癌患者的潜力。研究人员选择了舌苔中的50种微生物蛋白,通过使用ZJC队列进行训练和测试,建立了用于分类胃癌患者和非胃癌患者的机器学习模型,KEGG富集表明这50种蛋白质主要与核糖体相关,这提示了胃癌患者舌苔微生物蛋白合成的重要变化。
该模型在训练集和内部测试集中表现出较高的准确性、灵敏性和特异性,训练集中的AUC 为 0.96,内部测试集中AUC为0.92,并在独立验证队列(多中心队列)中表现出很好的性能(AUC = 0.87)。这些结果表明舌苔的微生物蛋白可以作为胃癌患者分类的有效生物标志物。
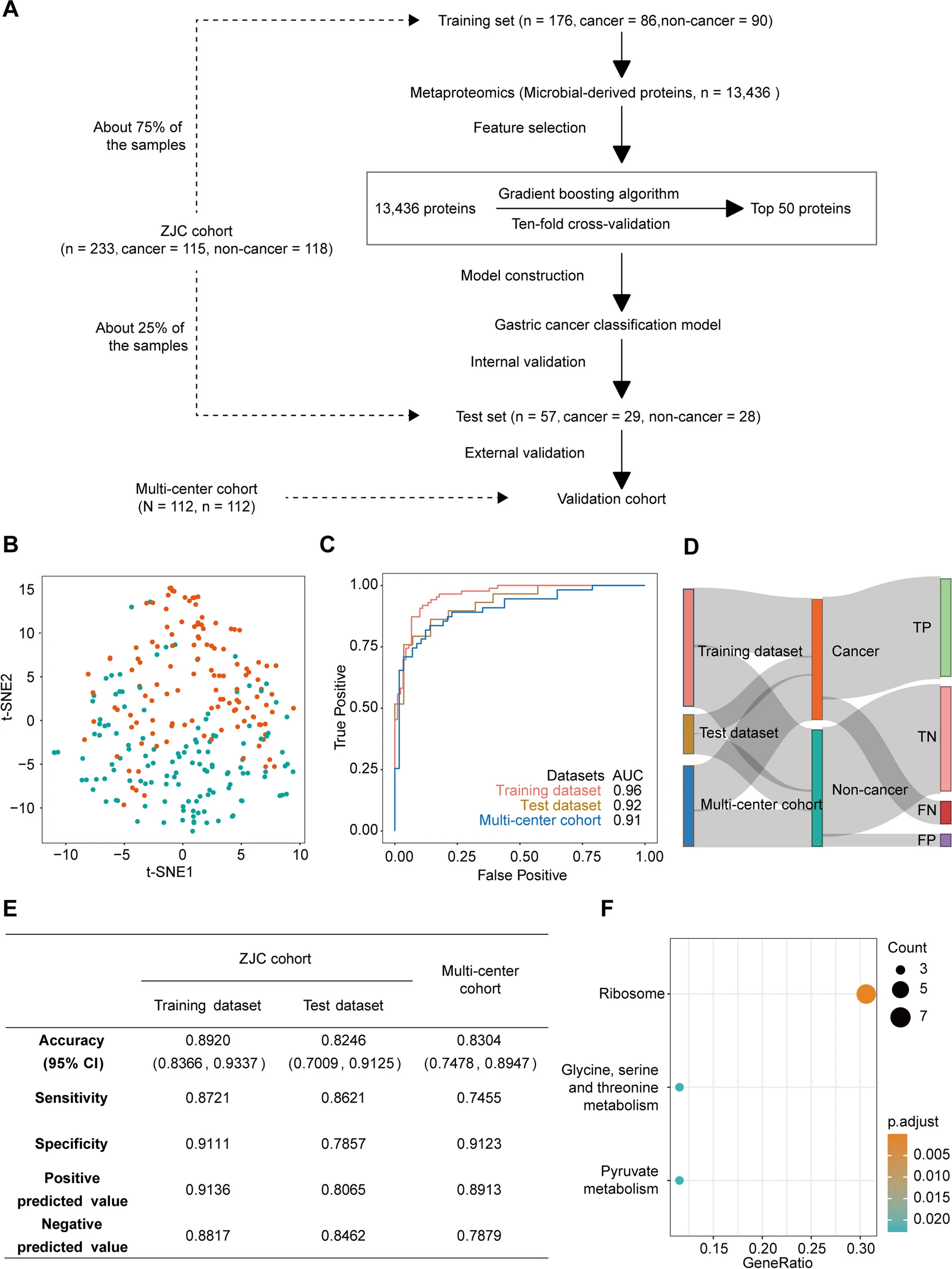
图5 基于微生物来源的舌苔蛋白的胃癌筛查模型
总 结
总体而言,该研究通过深入分析舌苔的蛋白质组学特征,揭示了胃癌患者舌苔中微生物和人源蛋白的变化,并成功构建了基于微生物蛋白的非侵入性胃癌筛查模型。这为发展新型的胃癌生物标志物和筛查方法提供了有力的支持。
文章链接:https://microbiomejournal.biomedcentral.com/articles/10.1186/s40168-023-01730-8
对 话 一 作
我们采访了文章一作(浙江省肿瘤医院的陈佳辉博士)以及共一作者(西湖实验室智能蛋白质组中心助理研究员孙莹莹博士),和他们聊了聊这篇有趣的研究。
Talk to 陈佳辉
Q 和团队在临床工作中遇到的胃癌临床痛点是什么?
A 还是早诊的问题。大多数早期胃癌的症状不典型或者几乎没有任何不适症状,导致很多患者发现时往往已经是晚期。我还在北大念博士的时候,就见到很多从各地尤其是医疗欠发达地区来的胃癌患者,有的用蛇皮袋背着全家的行囊来北京求医看病,但治疗效果不理想,究其原因是发现的时候已经太晚了。如果能用更为简单和精准的方式来早期诊断胃癌,(这些病人)就会有很好的治疗效果。
从博士期间我们就致力于开发一些胃癌早诊技术,包括以前我做的关于cfDNA的研究。回到杭州来到程向东老师团队,团队的特色之一就是从舌苔角度去研究胃癌,所以有机会通过舌苔来实现胃癌的早诊。就目前而言,全民胃镜检查(查胃癌)是不现实的,我们的目的是把胃癌高风险人群给筛选出来,然后再让他们去做胃镜检查。
Q 此次胃癌的研究方向为什么是舌苔蛋白组?
A 根据胃癌在舌苔图像上的表现,此前我们团队已经有了利用AI识别图像并构建模型的方式去识别个体患癌情况。传统中医上说望闻问切,望诊会看病人的舌苔,但这是第一步,后面为了探究里面的具体机制,我们还要从蛋白组的角度,从代谢、基因的角度去深入研究。我所在的浙江省肿瘤医院-国家癌症区域医疗中心针对胃癌进行有组织的科研攻关,我所参与的部分就是蛋白质(的角度)。
Q 这篇研究过程中遇到哪些困难?
A 最大的困难是舌苔(宏蛋白组)的参考方法不多,包括宏蛋白组的建库和整体方法的构建,西湖大学蛋白质组大数据实验室和西湖欧米的同事在这些工作中提供了很大的帮助。包括后期数据处理阶段,孙莹莹博士帮大家一起分析,最终我们得到了现在这些结果。
Q 相比AI舌象特征探索胃癌早诊,舌苔宏蛋白组研究有哪些优缺点?
A 舌象和舌苔蛋白组二者是从不同角度去研究胃癌。就图像而言,(AI模型)在医学上的可解释性会稍弱一些,包括中医上说胃癌患者舌苔可能厚一点或者腻一点,这些判断都需要更好的 “科学解释”。此次我们基于蛋白组学的优势,从蛋白质的角度去解释这个问题。我们和西湖大学蛋白质组大数据实验室合作,实验室DIA方法的蛋白质鉴定量非常高,尤其是微生物蛋白(1万多种),远远超出了现有的已公布的技术方法。如此庞大数量的蛋白质,有助于我们更好地去探索和 “解释” 舌苔与胃癌的联系。
宏蛋白组的蛋白鉴定量非常高,而且灵敏度很高。但对于早诊而言,鉴定到的信息太多了,不是所有信息都是我们想要的,所以目前它只是一个探索性的科研手段。其次,目前在实验室进行组学研究成本仍然较高,我们想开发既能大规模进行筛查、价格又相对较低的胃癌筛查技术。后期,我们在蛋白组学方向上可能通过尝试PRM(平行反应监测)、MRM(质谱多反应监测)等技术,争取把继续降低成本,服务于大众。
Talk to 孙莹莹
Q 过往对口腔/舌苔蛋白组的研究有很多,为什么这次不做蛋白组,而是 “宏蛋白组”?
A 以往口腔蛋白组仅测得的是人类蛋白,而宏蛋白组不仅可以测到人类蛋白,还可以测到群落中微生物的蛋白。而微生物正是我们这次重点研究的对象,通过测得微生物所表达的蛋白,来研究口腔微生物的功能及其与胃癌的关联。
Q 为什么对口腔宏蛋白质组学的研究仍处于初期阶段?
A 实际上相比于宏基因组、宏转录组,整个宏蛋白质组学的研究都处于相对初期的阶段,一方面是受限于质谱技术发展的限制,另一方面是宏蛋白质组学分析方法的瓶颈。不同于传统蛋白组学分析,宏蛋白组学分析时所采用的参考数据库非常大,通常是人类数据库的上百倍,需要更大的计算资源和更高效的算法。









