近日,剑桥大学医学院 Claudia Langenberg 团队在 Lancet Digital Health(IF:23.8)发表了新文章 Proteomic prediction of diverse incident diseases: a machine learning-guided biomarker discovery study using data from a prospective cohort study,探索了广谱蛋白质组学(Broad-capture proteomic)技术在多种疾病预测中的潜力。

图1 论文截图
- 提纲挈领 -
研究通过EPIC-Norfolk前瞻性队列研究,利用机器学习方法对2923种血清蛋白质进行分析,旨在评估蛋白质模型在24种疾病预测中的表现。研究发现,包含少量蛋白质(如五种蛋白质)的稀疏模型(sparse protein models)在17种疾病中优于多基因风险评分(PRS),并在7种疾病(如2型糖尿病和肺癌)的预测中显著提高了基本患者信息模型的表现(C指数提升范围为0.02至0.11)。此外,通过识别十种多病蛋白质,可以在多个疾病中实现较高的预测性能(中位C指数为0.72),表明这些蛋白质可能反映了共享的疾病机制,有潜力在临床应用中提供成本效益高的策略。
研究包括两个子群体:随机抽取的亚群(1759人)和在10年内发展出十种罕见疾病的群体(989人)。随机亚群的平均基线年龄为58.79岁,56.28%为女性;罕见疾病组的平均基线年龄为64.56岁,48.74%为女性。
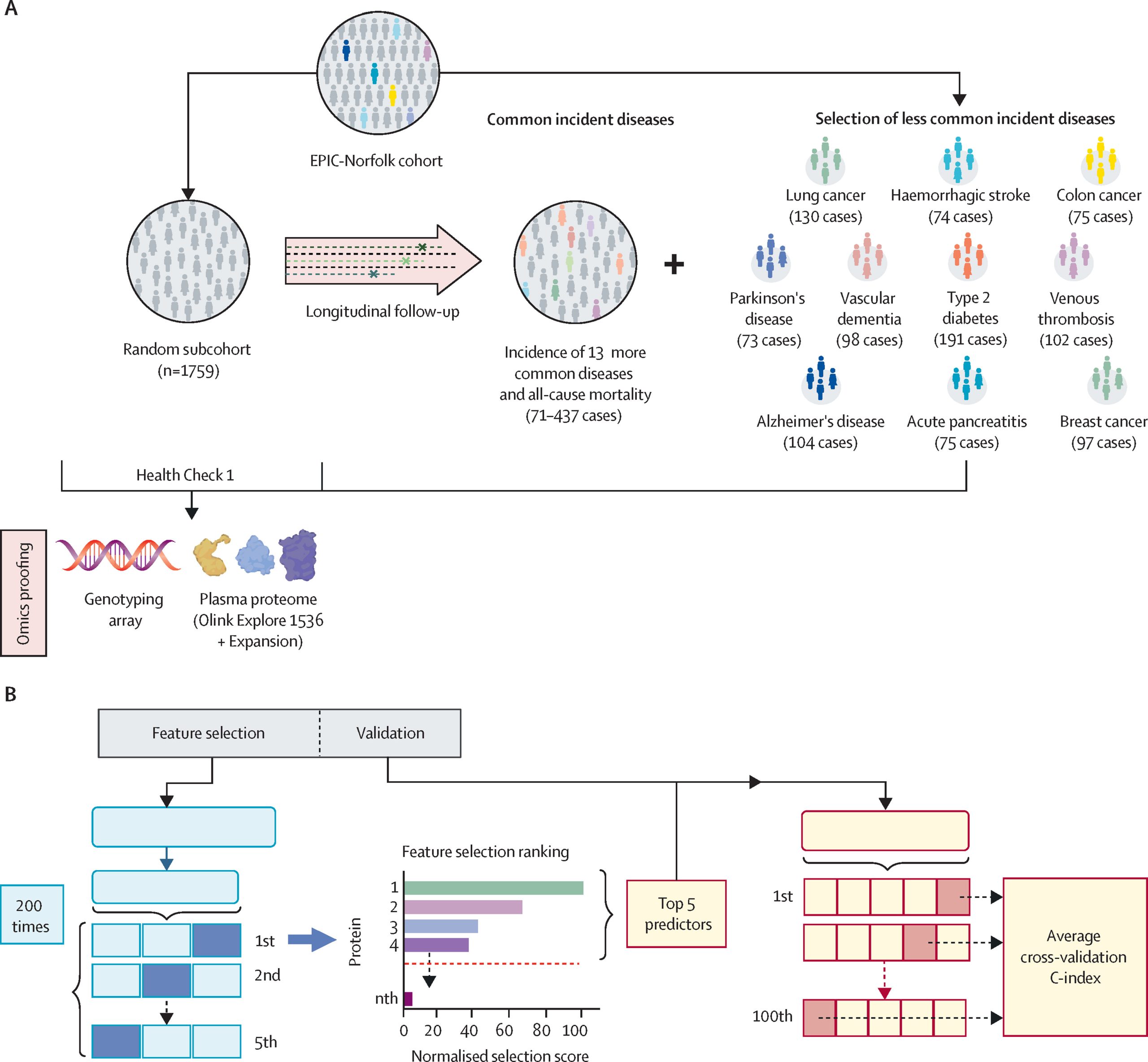
图2 研究设计
结果1:
蛋白质模型的预测性能
研究构建了24种不同结果的稀疏蛋白质模型,使用最少五种蛋白质的模型达到中位C指数0.67,比使用全部2319种蛋白质的模型表现更好,提升了0.04的中位C指数。
对于11种疾病,蛋白质模型的预测性能(中位C指数0.74)与基于患者信息的模型(中位C指数0.71)相当或更好。蛋白质模型在17种疾病中优于多基因风险评分(PRS),其中五种蛋白模型的C指数高出0.13。
结果2:
蛋白质和患者信息模型的结合
在七种疾病中,前面五种蛋白质加入到患者信息模型中后,预测性能显著提高,如2型糖尿病(C指数提高0.11)、前列腺癌(提高0.10)和全因过早死亡(提高0.08)(还包括COPD、肺癌、肾病和心力衰竭)。这些七种疾病的综合模型的中位C指数为0.82。
结果3:
多病蛋白质signature
研究还尝试建立一种可以预测多种疾病的单一稀疏蛋白质signature。前十种多病蛋白质在21种疾病中取得了中位C指数为0.72的预测性能,优于疾病特异性蛋白质signature。
这十种蛋白质在六种疾病和全因过早死亡的预测中提高了患者信息模型的预测性能(C指数提高0.02至0.06),中位C指数为0.81。
结果4:
关键预测蛋白质
研究发现了一些已知的临床生物标志物,以及一些在文献中很少提及但预测效果强的蛋白质,如CXCL17(用于肺癌和COPD)和LMOD1(用于肾病)。
在多种疾病中,26种蛋白质在两个或更多疾病中被共享,表明这些蛋白质可能指示了多种疾病的共享机制。
在设计探索性研究时,未发现蛋白质在特定疾病相关组(如心血管代谢、炎症、肿瘤学或神经学)中的富集现象。
总 结
总的来说,本研究展示了广谱蛋白质组学在多种疾病预测中的价值。通过机器学习框架,可以提取出稀疏的生物标志物panel,用于多种疾病的预测。这一发现为蛋白质组学在临床预测中的应用提供了新的方向,并有助于后续研究进一步验证和推广这些模型。
文章链接:
https://www.thelancet.com/journals/landig/article/PIIS2589-7500(24)00087-6/fulltext#%20








