文献目录
1 ARID1A在多发性骨髓瘤中的关键作用
2 泛癌蛋白质组学揭示新的治疗靶点
3 融合多组学数据和空间信息的高分辨率组织解析新方法
4 肺腺癌的N6-甲基腺苷表观转录组景观
5 蛋白质的预言:机器学习揭示多病预测的新视界
6 多发性骨髓瘤的蛋白质组学图谱
7 多组学揭示唐氏综合征的分子和免疫亚型
8 使用自监督深度学习对非标记定量质谱蛋白质组学数据进行归纳
一起来看看上周的蛋白质组学精选优质文献吧!
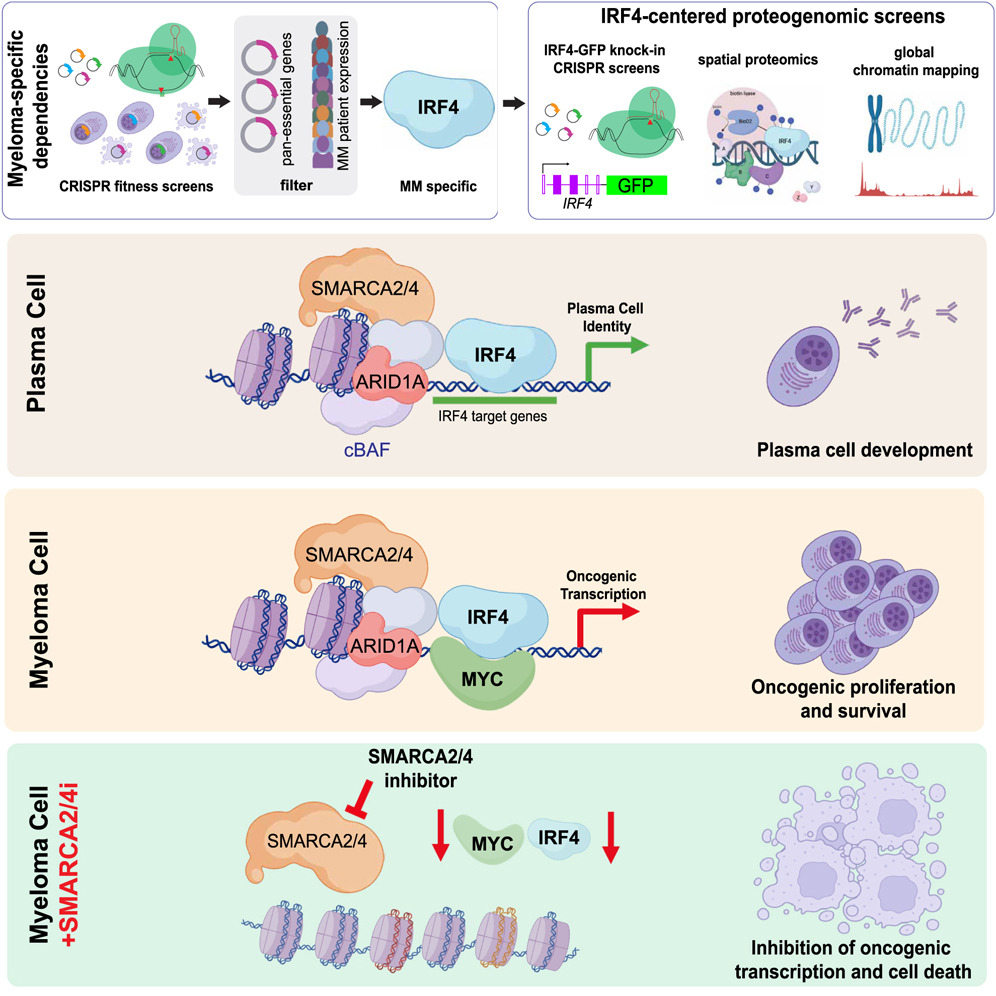
1.(Cancer Cell,48.8)ARID1A在多发性骨髓瘤中的关键作用

6月20日,美国国家癌症研究所的Ryan M. Young团队在 Cancer Cell 发表了多发性骨髓瘤的多组学研究。文章通过结合功能基因组筛选、空间蛋白质组学和全局染色质映射(global chromatin mapping)的多组学方法,揭示了ARID1A在多发性骨髓瘤(MM)中对IRF4表达和功能的关键作用。
研究发现,ARID1A通过SWI/SNF染色质重塑复合体促进IRF4表达,其缺失会破坏IRF4依赖的转录网络并阻碍浆细胞分化。靶向SWI/SNF活性可导致IRF4靶基因的快速表达丧失,降低MYC致癌基因表达,并对MM细胞产生显著毒性。蛋白质组学分析进一步支持了这些发现,显示出靶向SWI/SNF活性对MM细胞生存的影响。
特别是,SMARCA2/4抑制剂在IMiD耐药的MM细胞中仍有效,并与MEK抑制剂联合使用时表现出协同毒性。这些发现为复发/难治性MM患者提供了新的治疗策略。
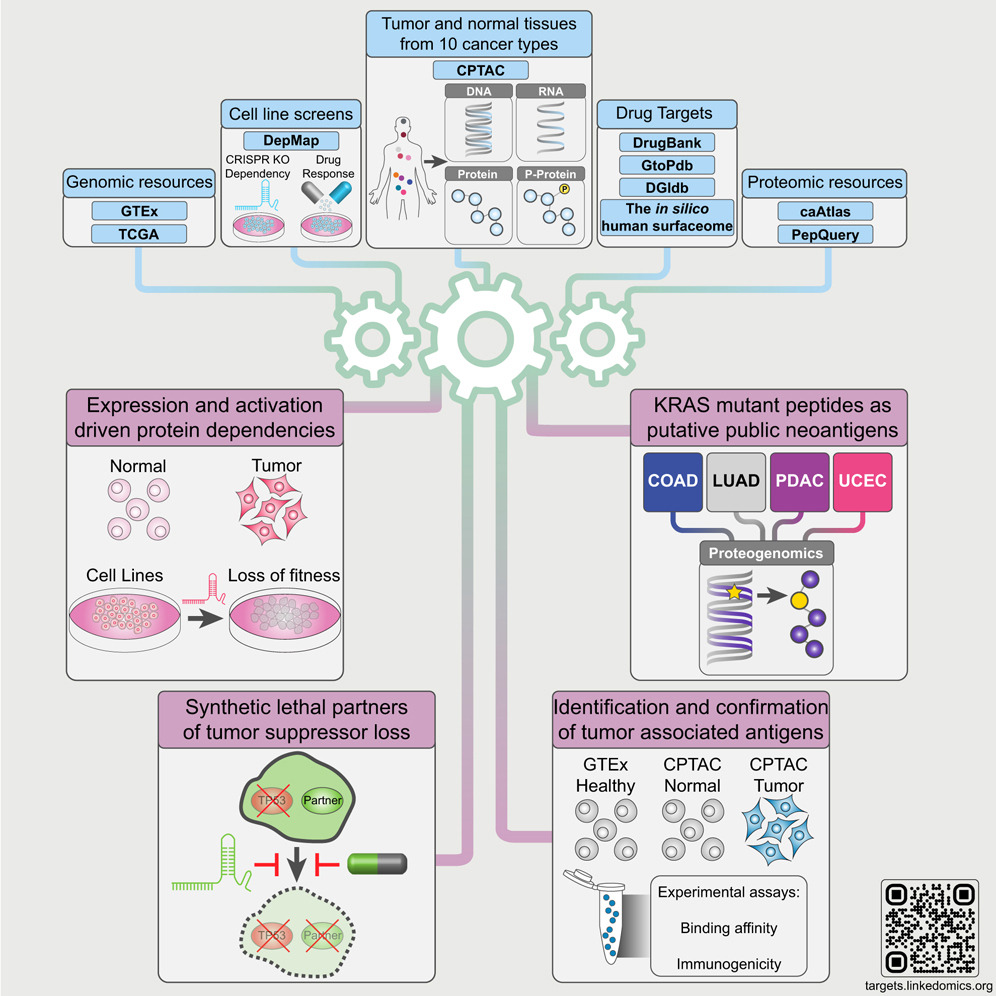
2.(Cell,IF:45.5)泛癌蛋白质组学揭示新的治疗靶点

6月24日,美国贝勒医学院章冰团队和复旦大学(附属中山医院)肝癌研究所高强团队在Cell上合作发文。通过对来自10种癌症类型的1,000多名患者的CPTAC蛋白质组学数据进行分析,提供了现有癌症药物靶点的见解,并扩展了治疗靶点的范围。
现有的癌症治疗药物仅靶向不到200种蛋白质,文章通过整合1,043名患者的CPTAC蛋白质组学数据和其他公共数据集,对10种癌症类型的2,863种可药物化蛋白质进行全癌症分析,揭示了蛋白质丰度范围及影响mRNA-蛋白质相关性的生物学因素。
结合蛋白质组学数据与细胞系基因筛选数据,研究人员识别出驱动癌症的蛋白质依赖性,并提出针对肿瘤抑制基因丧失的合成致死性策略。
研究还通过计算和实验确认,优先排序了KRAS突变肽段作为新抗原。这些分析结果在https://targets.linkedomics.org提供,用于伴随诊断、药物重新利用和治疗开发,为癌症治疗提供了重要资源。
3.(Nat Methods,IF: 36.1)融合多组学数据和空间信息的高分辨率组织解析新方法

6月21日,新加坡科技研究局(A*STAR)分子与细胞生物学研究院(IMCB)的研究人员与合作团队在 Nature Methods 发文,介绍了空间多组学数据分析工具SpatialGlue。
文章提出了一个名为 SpatialGlue 的图神经网络(GNN)模型,可通过整合蛋白质组学在内的多组学数据和空间信息更高分辨率地解析组织样本的空间域。通过一系列实验,SpatialGlue 展示了在模拟数据和实验数据上的优越性能,包括在人类淋巴结和小鼠脑组织等数据集上超越了现有的多种方法。
该模型通过双重注意力机制,在不同组学和空间信息之间进行有效的权重分配,从而准确地标记和分离不同的细胞类型和解剖结构。
实验结果表明,SpatialGlue 在多个评价指标上(如Moran’s I和Jaccard相似度)均优于其他方法,并在多组学数据整合和空间域解析中表现出更高的准确性和稳定性。
欧米解读:Nat Methods|SpatialGlue:融合多组学数据和空间信息的高分辨率组织解析新方法
4.(Cancer Discov,IF: 29.7)肺腺癌的N6-甲基腺苷表观转录组景观

6月25日,加拿大玛格丽特公主癌症中心的 Housheng Hansen. He 团队在 Cancer Discovery 发文,探索了肺腺癌的N6-甲基腺苷表观转录组景观。
对原发性肿瘤的全面m6A表观转录组分析仍然大部分未被探索。在此,研究人员对10个非肿瘤性肺组织(NL)和51个肺腺癌肿瘤(LUAD)的m6A表观转录组进行了分析,并整合了相应的转录组、蛋白质组和广泛的临床注释。
研究人员识别出通过m6A修饰与疾病进展独特相关的基因和簇。他们发现,与NL组织相比,肿瘤中有430个转录本呈低甲基化,222个呈高甲基化。在这些基因中,EML4作为一个新的转移驱动因子,显示出在肿瘤中显著的高甲基化。
此外,m6A修饰促进了EML4的翻译,导致其在原发性肿瘤中的广泛过表达。在功能上,EML4通过与ARPC1A相互作用,调节细胞骨架动态,增强板状伪足形成、细胞运动、局部侵袭和转移。在临床上,高EML4蛋白丰度与转移特征相关。METTL3小分子抑制剂显著减少了EML4的m6A和蛋白丰度,并有效抑制了体内肺转移。
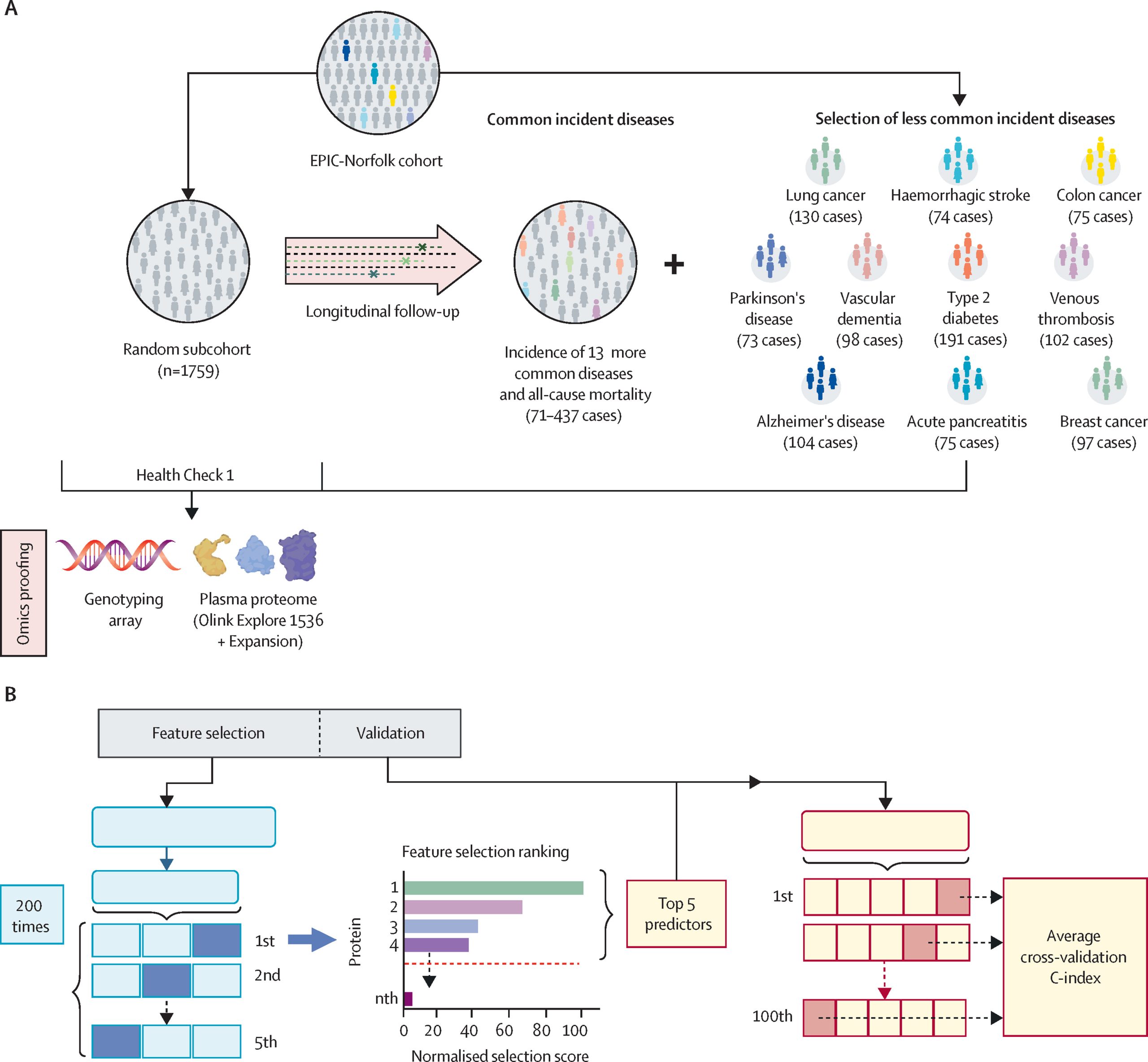
5.(Lancet Digit Health,IF: 23.8)蛋白质的预言:机器学习揭示多病预测的新视界

近日,剑桥大学医学院 Claudia Langenberg 团队在 Lancet Digital Health 发文,探索了广谱蛋白质组学(Broad-capture proteomic)技术在多种疾病预测中的潜力。
研究通过EPIC-Norfolk前瞻性队列研究,利用机器学习方法对2923种血清蛋白质进行分析,旨在评估蛋白质模型在24种疾病预测中的表现。
研究发现,包含少量蛋白质(如五种蛋白质)的稀疏模型(sparse protein models)在17种疾病中优于多基因风险评分(PRS),并在7种疾病(如2型糖尿病和肺癌)的预测中显著提高了基本患者信息模型的表现(C指数提升范围为0.02至0.11)。
此外,通过识别十种多病蛋白质,可以在多个疾病中实现较高的预测性能(中位C指数为0.72),表明这些蛋白质可能反映了共享的疾病机制,有潜力在临床应用中提供成本效益高的策略。
6.(Nat Cancer,IF: 23.5)多发性骨髓瘤的蛋白质组学图谱

6月28日,柏林夏里特医学院、德国马克斯·德尔布吕克分子医学中心以及维尔茨堡大学医院的研究团队合作在 Nature Cancer 发表了多发性骨髓瘤多组学新研究,揭示了蛋白质组与发性骨髓瘤疾病进展和预后的关联,并识别出潜在治疗靶点。
研究人员对138例不同病程阶段的多发性骨髓瘤患者样本进行多组学分析,包括TMT定量(磷酸化)蛋白质组学、RNA测序和纳米孔DNA测序,发现了恶性浆细胞相较于健康浆细胞在蛋白质组水平上的显著失调,受到染色体改变和转录后调控的影响。
研究识别出了一种与侵袭性疾病相关的预后蛋白质signature,并揭示了多种遗传亚型特异的失调蛋白和路径,提供了潜在的治疗靶点。这项研究展示了蛋白质基因组学在多发性骨髓瘤研究中的潜力,为改善疾病风险分层和开发新疗法提供了重要资源。
7.(Nat Comm,IF: 14.7)多组学揭示唐氏综合征的分子和免疫亚型

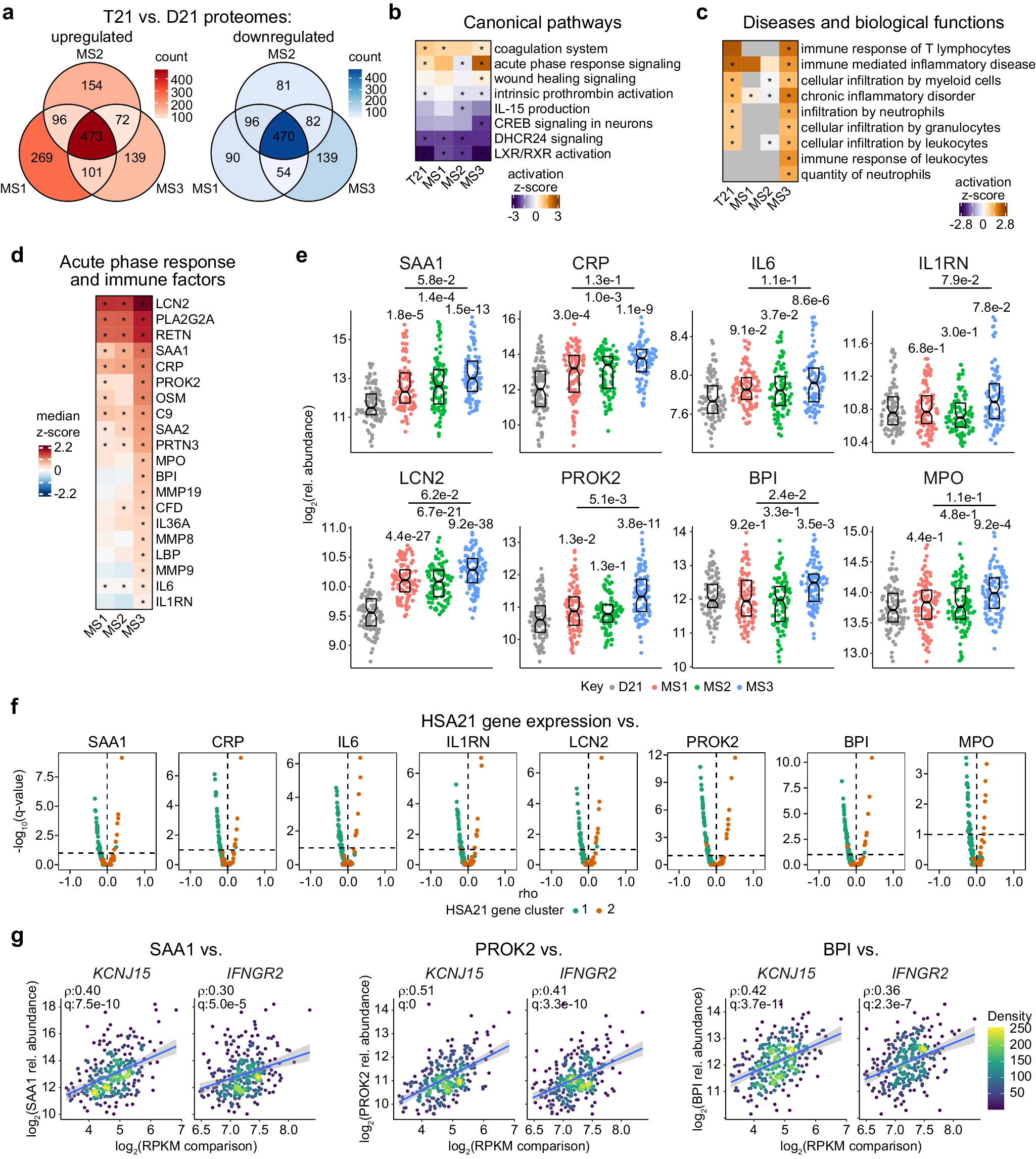
6月28日,美国科罗拉多大学医学院 Linda Crnic 唐氏综合症研究所的研究人员在 Nature Communications 发文,通过分析白血细胞转录组、血浆蛋白组和代谢组以及免疫细胞分布,揭示了唐氏综合征患者分子和免疫亚型的多样性。
通过分析大量唐氏综合征患者的白血细胞转录组,研究人员发现了21号染色体基因过表达存在显著的个体间变异。研究揭示了两个主要的基因簇及三种分子亚型,这些亚型在炎症、免疫反应、细胞增长与代谢等关键病理过程上表现出明显的差异。
通过全面的多组学分析,研究不仅深化了对唐氏综合征分子异质性的理解,还为将来个性化医疗策略的开发提供了重要基础。
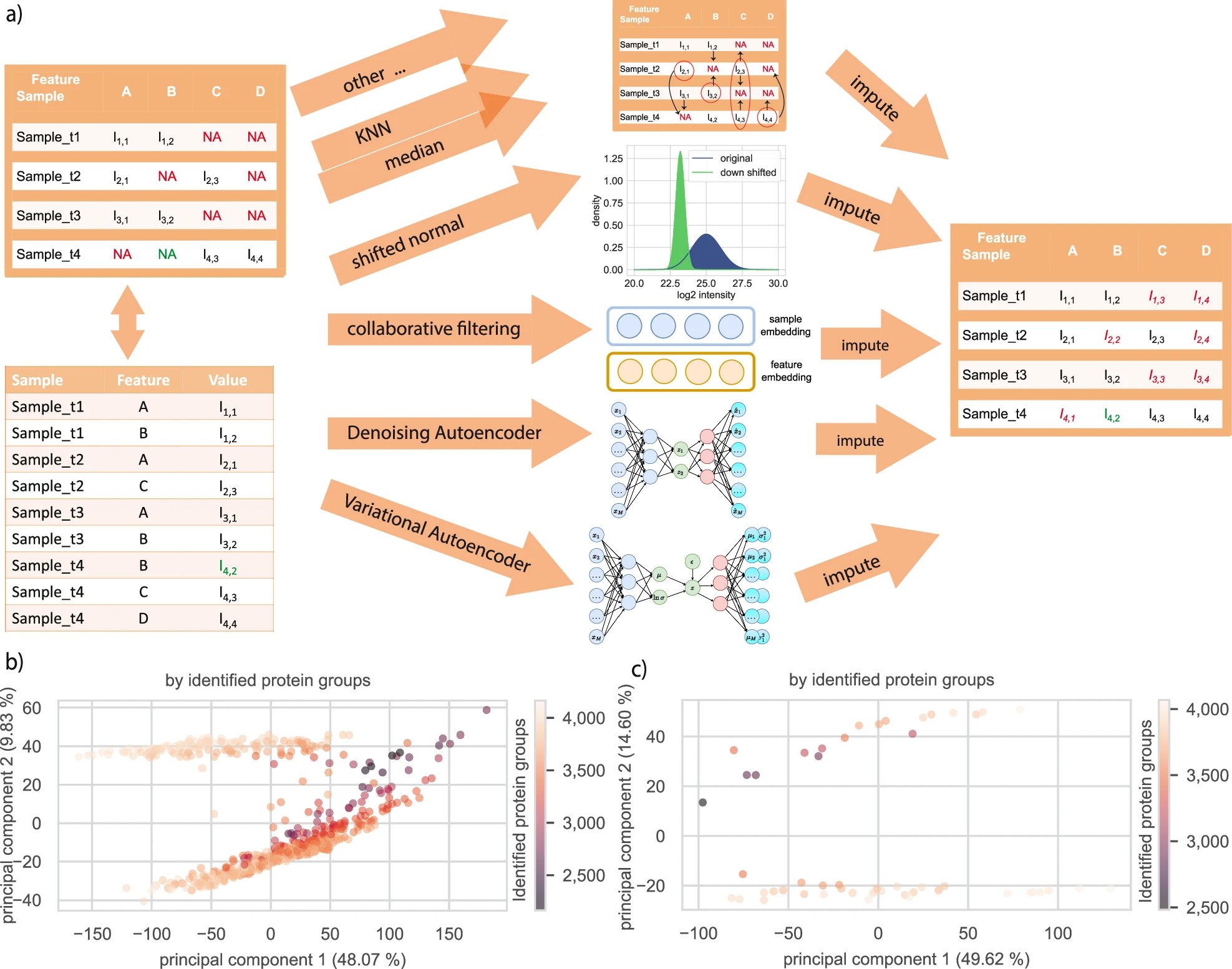
8.(Nat Comm,IF: 14.7)使用自监督深度学习对非标记定量质谱蛋白质组学数据进行归纳

6月26日,哥本哈根大学的 Simon Rasmussen 及 Lars Juhl Jensen 团队在 Nature Communications 发文,讨论了如何使用深度学习方法对质谱蛋白质组学数据进行缺失值插补。
文章介绍了一种名为PIMMS(proteomics imputation modeling mass spectrometry,蛋白质组学插补建模质谱)的新方法,使用协同过滤(CF)、去噪自编码器(DAE)和变分自编码器(VAE)等自监督深度学习模型对非标记定量(LFQ)质谱蛋白质组学数据进行缺失值插补。
在358名酒精性肝病(ALD)患者的血浆蛋白质组数据中,移除20%的强度后,PIMMS-VAE成功恢复了15个显著丰度的蛋白质组中的17个。分析完整数据集时,研究人员使用PIMMS-VAE比未插补多鉴定出30个(增加13.2%)显著差异丰度的蛋白质,并发现这些蛋白质能预测ALD的进展,表明深度学习方法在处理大规模蛋白质组学数据中的潜力。