多数药物通过作用于蛋白质来实现疗效,它们可导致蛋白质的生成或降解,或通过其他方式来调控蛋白质使药物发挥疗效。药物调控的蛋白质翻译后修饰(Post-translational modifications,PTMs)可作为识别药物调节途径的分子标识,阐明受药物干扰后细胞下游表型响应,这对于药物作用机制的研究有着重要作用。
以此为研究逻辑,德国慕尼黑工业大学生命科学学院分子生命科学系Bernhard Kuster教授在science上发表了文章“Decrypting drug actions and protein modifications by dose- and time-resolved proteomics”。

在文章中,作者提出了一种定量蛋白质组学方法,名为DecryptM,通过系统地测量药物剂量和时间对细胞中的PTMs(磷酸化、乙酰化、泛素化)的干扰,用于评估多种癌症药物的靶点和相关通路,以及药物作用机制(Mechanism of action,MoA)(图1)。文章中讨论了六个药物类别的例子,说明了DecryptM分析如何用于表征细胞中药物的 MoA,并为 PTMs分配功能。作者选择了13个人类癌细胞系,用31种抗癌药物进行了DecryptM分析,量化了细胞中数千个PTM事件的药物调节,分析结果表明了DecryptM这一方法有广泛的适用性。实验数据也被整理到蛋白质组学数据库ProteomicsDB中,以供参考和探索。
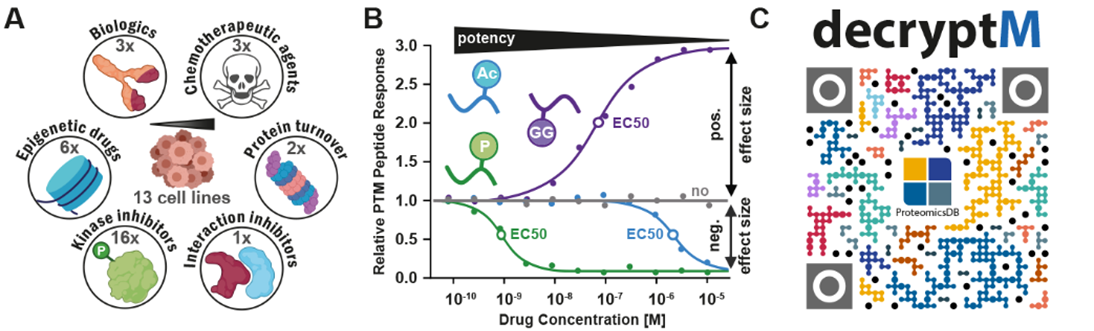
图1 研究所用药物及细胞系数量、涉及的PTMs类型以及ProteomicsDB二维码链接
与传统的只在一个药物浓度下对药物效应大小进行复制测量的差异分析相比,DecryptM方法让细胞经过不断增加浓度的药物处理,得到剂量-反应曲线,提供EC50值,效应大小和额外的曲线拟合参数。通过使用DecryptM方法分析A431表皮癌细胞的磷酸化蛋白质组在EGFR抑制剂阿法替尼、吉非替尼和多激酶抑制剂达沙替尼的作用下的情况,作者发现剂量-反应曲线独特地说明了药效大小,并且大多数经调节的磷酸化肽段以及它们所涉及的通路与其靶点的亲和力密切相关(图2)。而药物靶点的亲和力(pKD)和细胞内药物诱导的 PTM 效应(pEC50)之间的密切关联,为DecryptM提供了潜在应用。
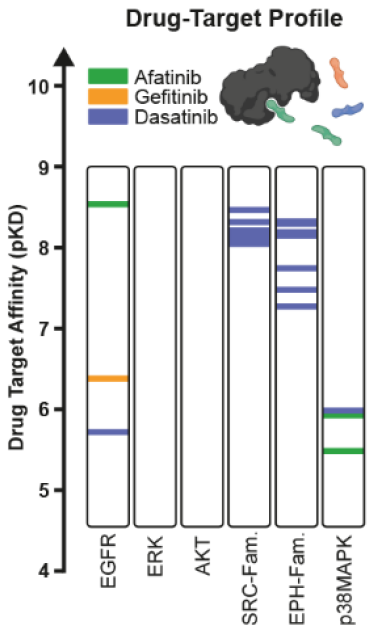
图2 药物-靶点亲和力测定结果
化学治疗是癌症治疗过程中极其重要的一个阶段。作者在K562慢性骨髓性白血病细胞中,为化疗药物微管稳定剂紫杉醇和抗代谢物阿糖胞苷和甲氨蝶呤进行了DecryptM分析。实验结果表明,在药物处理30分钟内,可以观察到与药物相关的磷酸肽被调节。通过对紫杉醇进行进一步分析,发现被上调的磷酸肽所对应的蛋白质都在微管稳定中发挥作用,说明了药物调节可以通过剂量依赖的测量获得极高的特异性,紫杉醇的分子机制也可以在30分钟后就被检测到(图3)。
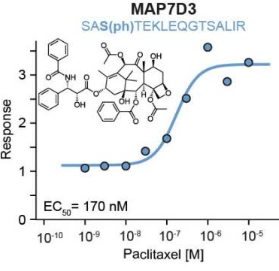
图3 紫杉醇处理细胞后蛋白磷酸肽表达情况
SHP099作为一种蛋白质相互作用抑制剂,是非受体酪氨酸磷酸酶SHP-2(PTPN11)的变构抑制剂,通过稳定SHP-2的非活性形式,从而阻断受体酪氨酸激酶(RTK)激活的MAPK途径,抑制癌细胞的生长。
为了验证这一机制,作者做了EGFR过表达的食管鳞状细胞癌细胞系KYSE-520中SHP099的30分钟DecryptM分析。分析结果不仅反映了这一机制,还表明GFR底物GAB1的磷酸化以剂量依赖的方式减少,许多转录调控因子在内的约30种其他蛋白质也显示出了调节作用。这些结果说明了药物学上的SHP-2失活如何导致致癌信号和转录活性的快速崩解。
通过在多发性骨髓瘤细胞RPMI 8226中对硼替佐米(Bortezomib,BTZ)和卡非佐米(Carfilzomib,CFZ)进行二维decryptM分析,进一步说明了该方法在表征MoA中的适用性。实验结果表明,这两种药物在该细胞中有相似的效果(图4),随着时间的推移,受到调节的磷酸化位点在不断增加,药效也更加强烈,与应激反应有关的转录因子和分子伴侣蛋白的表达发生了变化。这表明随着时间的推移,功能性蛋白酶体的数量在不断减少。
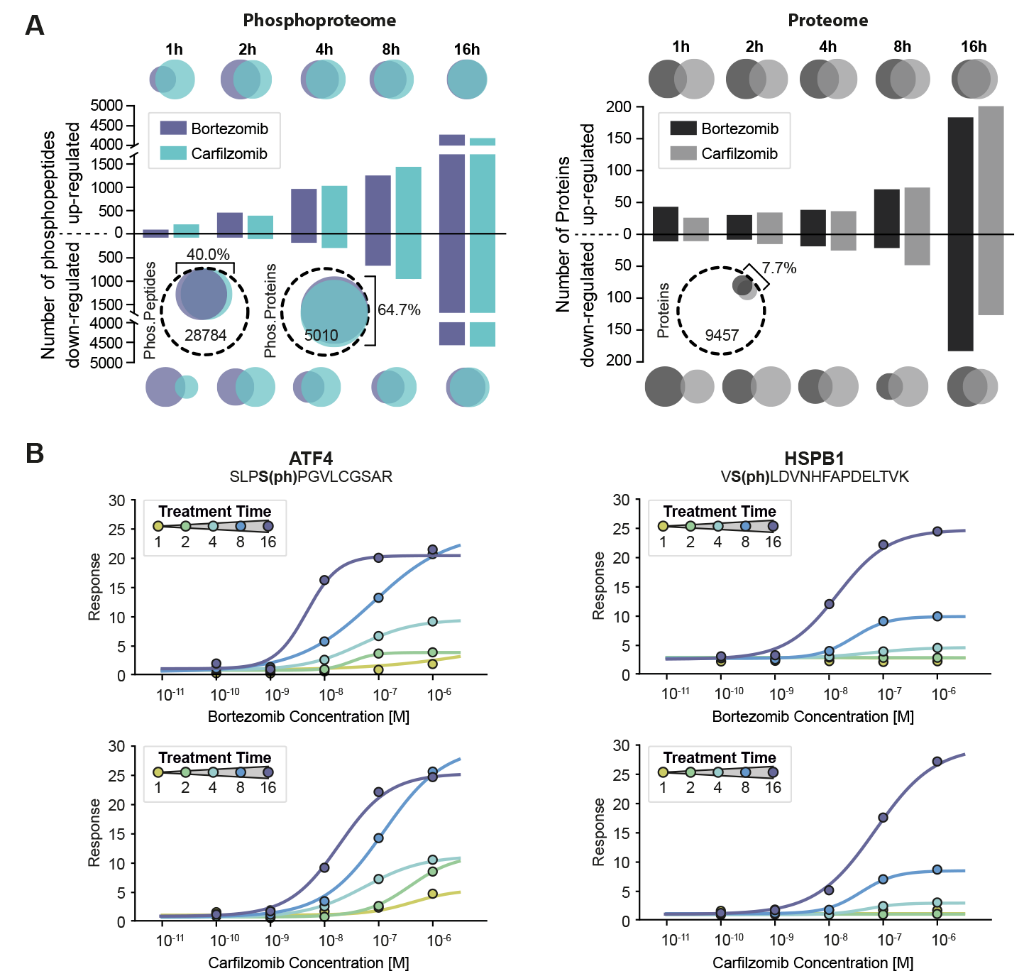
图4 BTZ和CFZ在RPMI 8226药效情况
为了进一步了解赖氨酸乙酰转移酶(KATs)和赖氨酸脱乙酰酶(KDACs)在细胞中的作用机制,作者选择将decryptM方法扩展到HeLa细胞的蛋白质乙酰化上。结果显示,双重多激酶和HDAC1/2/3抑制剂CUDC-101可上调139个乙酰化肽,潜在性弱于磷酸化肽(图5);HDAC抑制剂沃利诺斯他在4小时之内并没有明显影响蛋白质磷酸化;天然化合物罗米地平在16小时后触发了磷酸化蛋白组的广泛和强烈的反应。结果证明药物的作用与其靶向选择性是一致的。
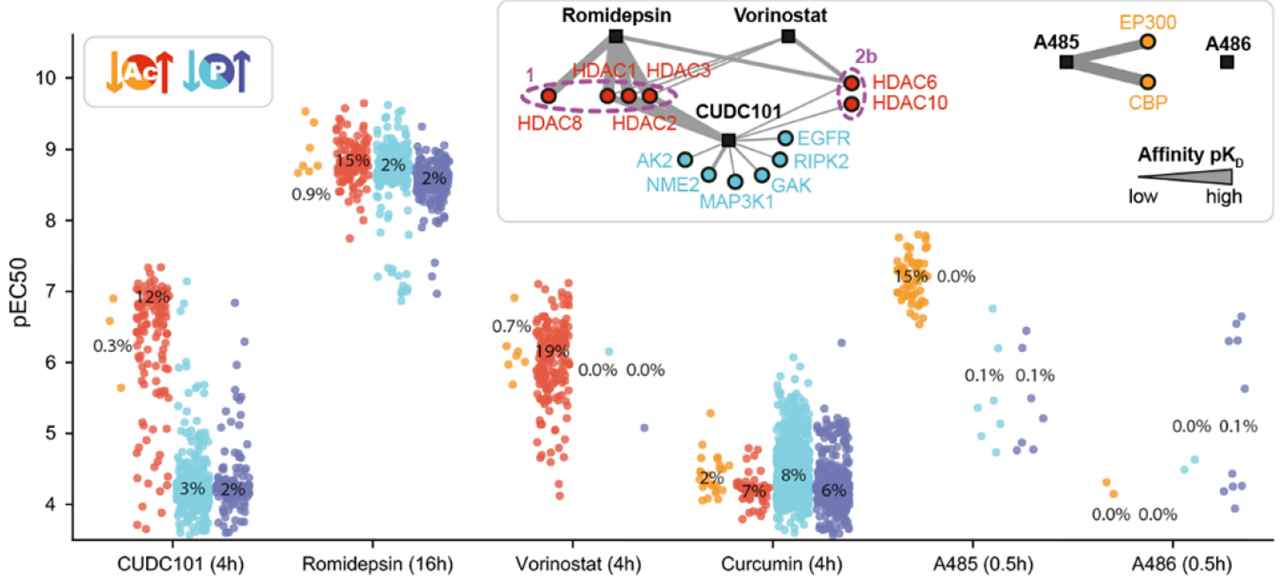
图5 细胞效价(pEC50)的药物调节的乙酰化(红色)和磷酸化肽段(蓝色)的条形图
激酶抑制剂(KI)作为肿瘤学中的重要药物,在细胞中影响那些PTM仍然知之甚少。为了系统地解决这个问题,作者在A549肺癌细胞中对10种具有不同靶点谱的KIs进行了decryptM分析,揭示了药物调控的磷酸化肽段(图6)以及蛋白质的结构域和无序区域。
实验结果显示,泛激酶抑制剂链霉素调节了近2500个磷酸化肽段,而其它药物调节的磷酸肽较少。许多KI的pEC50分布较窄但呈双峰分布,表明在非常高的浓度下,不同的靶标和途径具有不同的效力或非特异性的细胞毒性。
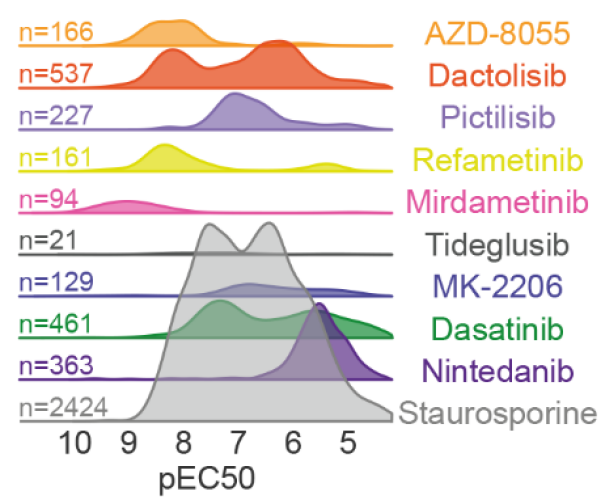
图6 受到药物调控的磷酸化肽段影响分布图
利妥昔单抗(Rituximab,RTX)被用于治疗B细胞恶性肿瘤,通过靶向MS4A1(CD20)。为了研究RTX的作用机制在多大程度上涉及磷酸化依赖的信号通路,作者选择了对RTX敏感的细胞系SU-DHL-4(B细胞淋巴瘤)和Ramos(Burkitt淋巴瘤)以及对RTX不敏感的浆细胞白血病细胞系ARH-77进行decryptM分析(图7)。分析结果显示,所有细胞系都快速结合了RTX,但对RTX敏感的细胞系调节了大量磷酸肽(图8)。在检测期间,B细胞受体(BCR)信号特征持续存在,但在2-24小时中,出现了细胞老化的信号和凋亡的信号,反映了细胞应激反应的增加和向凋亡细胞死亡的转变。实验结果证明了RTX通过过度激活BCR杀死CD20阳性的B细胞。
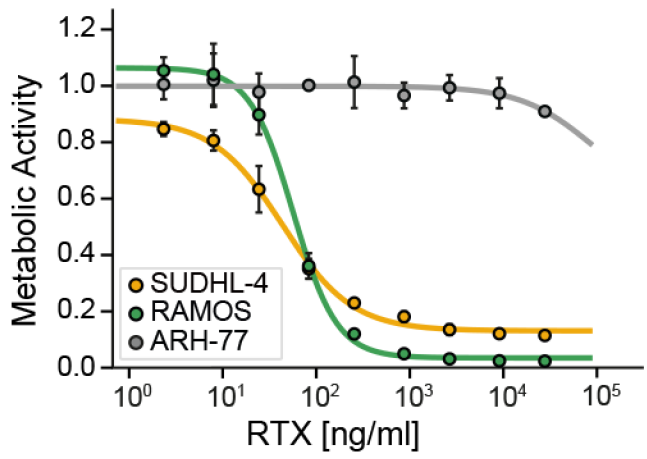
图7 利妥昔单抗敏感和耐药细胞系的存活率试验
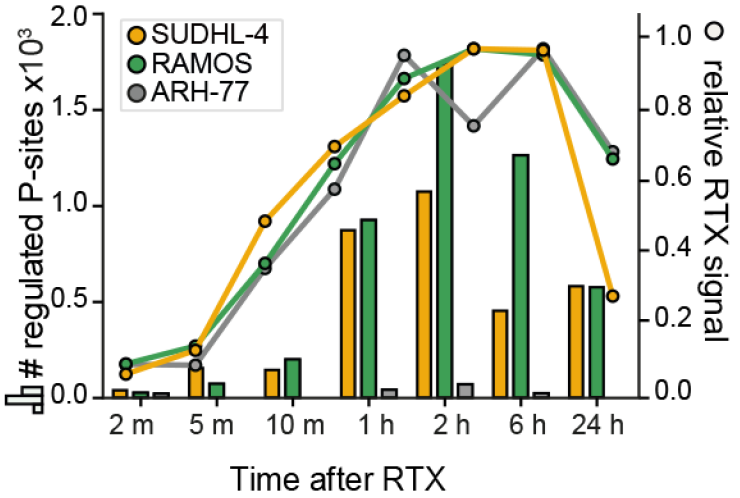
图8 RTX处理的不同时间点对同一细胞系的剂量反应调节的磷酸化肽段数目和利妥昔单抗与这些细胞结合的动力学
通过上述例子说明,decryptM在表征药物的MoA、产生药物特异性PTM特征、研究耐药性机制以及将意义不明确的药物调节的PTM位点置于功能环境中的巨大潜力,可应用于任何通过影响翻译后修饰或蛋白质表达调节细胞活性的分子,包括 GPCR 配体、细胞因子、趋化因子、辅因子、代谢产物、生物制剂、肽类或激素等等。
原文链接:https://www.science.org/doi/10.1126/science.ade3925
文章解读:于洁
审校:张程
多数药物通过作用于蛋白质来实现疗效,它们可导致蛋白质的生成或降解,或通过其他方式来调控蛋白质使药物发挥疗效。药物调控的蛋白质翻译后修饰(Post-translational modifications,PTMs)可作为识别药物调节途径的分子标识,阐明受药物干扰后细胞下游表型响应,这对于药物作用机制的研究有着重要作用。
以此为研究逻辑,德国慕尼黑工业大学生命科学学院分子生命科学系Bernhard Kuster教授在science上发表了文章“Decrypting drug actions and protein modifications by dose- and time-resolved proteomics”。

在文章中,作者提出了一种定量蛋白质组学方法,名为DecryptM,通过系统地测量药物剂量和时间对细胞中的PTMs(磷酸化、乙酰化、泛素化)的干扰,用于评估多种癌症药物的靶点和相关通路,以及药物作用机制(Mechanism of action,MoA)(图1)。文章中讨论了六个药物类别的例子,说明了DecryptM分析如何用于表征细胞中药物的 MoA,并为 PTMs分配功能。作者选择了13个人类癌细胞系,用31种抗癌药物进行了DecryptM分析,量化了细胞中数千个PTM事件的药物调节,分析结果表明了DecryptM这一方法有广泛的适用性。实验数据也被整理到蛋白质组学数据库ProteomicsDB中,以供参考和探索。

图1 研究所用药物及细胞系数量、涉及的PTMs类型以及ProteomicsDB二维码链接
与传统的只在一个药物浓度下对药物效应大小进行复制测量的差异分析相比,DecryptM方法让细胞经过不断增加浓度的药物处理,得到剂量-反应曲线,提供EC50值,效应大小和额外的曲线拟合参数。通过使用DecryptM方法分析A431表皮癌细胞的磷酸化蛋白质组在EGFR抑制剂阿法替尼、吉非替尼和多激酶抑制剂达沙替尼的作用下的情况,作者发现剂量-反应曲线独特地说明了药效大小,并且大多数经调节的磷酸化肽段以及它们所涉及的通路与其靶点的亲和力密切相关(图2)。而药物靶点的亲和力(pKD)和细胞内药物诱导的 PTM 效应(pEC50)之间的密切关联,为DecryptM提供了潜在应用。

图2 药物-靶点亲和力测定结果
化学治疗是癌症治疗过程中极其重要的一个阶段。作者在K562慢性骨髓性白血病细胞中,为化疗药物微管稳定剂紫杉醇和抗代谢物阿糖胞苷和甲氨蝶呤进行了DecryptM分析。实验结果表明,在药物处理30分钟内,可以观察到与药物相关的磷酸肽被调节。通过对紫杉醇进行进一步分析,发现被上调的磷酸肽所对应的蛋白质都在微管稳定中发挥作用,说明了药物调节可以通过剂量依赖的测量获得极高的特异性,紫杉醇的分子机制也可以在30分钟后就被检测到(图3)。

图3 紫杉醇处理细胞后蛋白磷酸肽表达情况
SHP099作为一种蛋白质相互作用抑制剂,是非受体酪氨酸磷酸酶SHP-2(PTPN11)的变构抑制剂,通过稳定SHP-2的非活性形式,从而阻断受体酪氨酸激酶(RTK)激活的MAPK途径,抑制癌细胞的生长。
为了验证这一机制,作者做了EGFR过表达的食管鳞状细胞癌细胞系KYSE-520中SHP099的30分钟DecryptM分析。分析结果不仅反映了这一机制,还表明GFR底物GAB1的磷酸化以剂量依赖的方式减少,许多转录调控因子在内的约30种其他蛋白质也显示出了调节作用。这些结果说明了药物学上的SHP-2失活如何导致致癌信号和转录活性的快速崩解。
通过在多发性骨髓瘤细胞RPMI 8226中对硼替佐米(Bortezomib,BTZ)和卡非佐米(Carfilzomib,CFZ)进行二维decryptM分析,进一步说明了该方法在表征MoA中的适用性。实验结果表明,这两种药物在该细胞中有相似的效果(图4),随着时间的推移,受到调节的磷酸化位点在不断增加,药效也更加强烈,与应激反应有关的转录因子和分子伴侣蛋白的表达发生了变化。这表明随着时间的推移,功能性蛋白酶体的数量在不断减少。

图4 BTZ和CFZ在RPMI 8226药效情况
为了进一步了解赖氨酸乙酰转移酶(KATs)和赖氨酸脱乙酰酶(KDACs)在细胞中的作用机制,作者选择将decryptM方法扩展到HeLa细胞的蛋白质乙酰化上。结果显示,双重多激酶和HDAC1/2/3抑制剂CUDC-101可上调139个乙酰化肽,潜在性弱于磷酸化肽(图5);HDAC抑制剂沃利诺斯他在4小时之内并没有明显影响蛋白质磷酸化;天然化合物罗米地平在16小时后触发了磷酸化蛋白组的广泛和强烈的反应。结果证明药物的作用与其靶向选择性是一致的。

图5 细胞效价(pEC50)的药物调节的乙酰化(红色)和磷酸化肽段(蓝色)的条形图
激酶抑制剂(KI)作为肿瘤学中的重要药物,在细胞中影响那些PTM仍然知之甚少。为了系统地解决这个问题,作者在A549肺癌细胞中对10种具有不同靶点谱的KIs进行了decryptM分析,揭示了药物调控的磷酸化肽段(图6)以及蛋白质的结构域和无序区域。
实验结果显示,泛激酶抑制剂链霉素调节了近2500个磷酸化肽段,而其它药物调节的磷酸肽较少。许多KI的pEC50分布较窄但呈双峰分布,表明在非常高的浓度下,不同的靶标和途径具有不同的效力或非特异性的细胞毒性。

图6 受到药物调控的磷酸化肽段影响分布图
利妥昔单抗(Rituximab,RTX)被用于治疗B细胞恶性肿瘤,通过靶向MS4A1(CD20)。为了研究RTX的作用机制在多大程度上涉及磷酸化依赖的信号通路,作者选择了对RTX敏感的细胞系SU-DHL-4(B细胞淋巴瘤)和Ramos(Burkitt淋巴瘤)以及对RTX不敏感的浆细胞白血病细胞系ARH-77进行decryptM分析(图7)。分析结果显示,所有细胞系都快速结合了RTX,但对RTX敏感的细胞系调节了大量磷酸肽(图8)。在检测期间,B细胞受体(BCR)信号特征持续存在,但在2-24小时中,出现了细胞老化的信号和凋亡的信号,反映了细胞应激反应的增加和向凋亡细胞死亡的转变。实验结果证明了RTX通过过度激活BCR杀死CD20阳性的B细胞。

图7 利妥昔单抗敏感和耐药细胞系的存活率试验

图8 RTX处理的不同时间点对同一细胞系的剂量反应调节的磷酸化肽段数目和利妥昔单抗与这些细胞结合的动力学
通过上述例子说明,decryptM在表征药物的MoA、产生药物特异性PTM特征、研究耐药性机制以及将意义不明确的药物调节的PTM位点置于功能环境中的巨大潜力,可应用于任何通过影响翻译后修饰或蛋白质表达调节细胞活性的分子,包括 GPCR 配体、细胞因子、趋化因子、辅因子、代谢产物、生物制剂、肽类或激素等等。
原文链接:https://www.science.org/doi/10.1126/science.ade3925
文章解读:于洁
审校:张程









