
文献目录
1 (Nature) Mann团队空间蛋白组学研究发现致命皮肤病治疗药物
2 (Nat Chem) 通过凝聚增强光交联进行时空蛋白质相互作用组分析
3 (Eur J Heart Fail) 蛋白质生物标志物预测心力衰竭进展的多队列研究
4 (Angew Chem Int Ed Engl) TEAQ:通过DIA-MS发现并筛选临床级生物标志物
5 (Nat Comm) FAXP:石蜡切片中单个亚细胞器的深度蛋白质组分析
6 (Microbiome) 宏蛋白组学揭示克罗恩病患者饮食干预的肠道微生物组功能变化
7 (Gut Microbes) 多组学研究揭示运动改变肠道真菌群促进代谢健康
8 (Cell Genom) AI赋能的微扰蛋白质组学:探索复杂生物系统的PMMP流程
一起来看看上周的蛋白质组学精选优质文献吧!
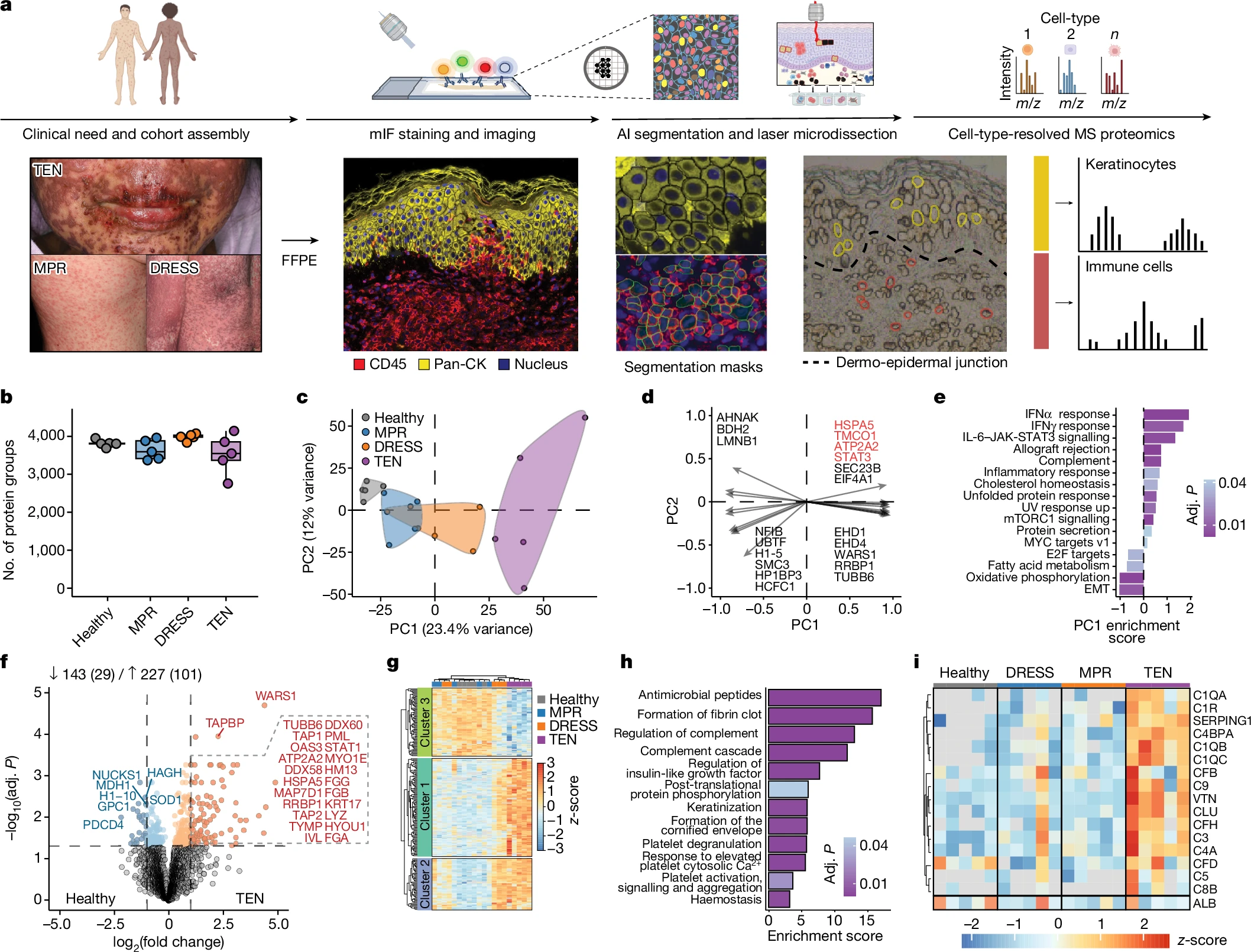
1.(Nature,IF:50.5)Mann团队空间蛋白组学研究发现致命皮肤病治疗药物

10月16日,马克斯·普朗克生物化学研究所 Matthias Mann 团队,联合慕尼黑大学医院、福建医科大学第一附属医院等单位,在 Nature 共同发表了中毒性表皮坏死松解症(TEN)的最新研究。通过深度视觉蛋白质组学(DVP)揭示了JAK/STAT信号通路在中毒性表皮坏死松解症中的关键作用,并验证了JAK抑制剂作为潜在疗法的有效性。
研究人员利用深度视觉蛋白质组学技术,对中毒性表皮坏死松解症患者的皮肤活检样本进行分析,定量了超过5000种蛋白质,揭示了JAK/STAT信号通路在TEN发病中的关键作用,尤其是干扰素信号和STAT1的磷酸化激活。
研究通过体外和体内模型验证了JAK抑制剂(如tofacitinib、baricitinib、abrocitinib和upadacitinib)能够有效降低角质形成细胞的细胞毒性,减少皮肤损伤,并加速上皮恢复。在病例研究中,7位TEN患者接受JAK抑制剂治疗后,均显示出显著的临床改善,表皮再生迅速且无副作用,表明JAK抑制剂有望成为TEN的有效疗法。
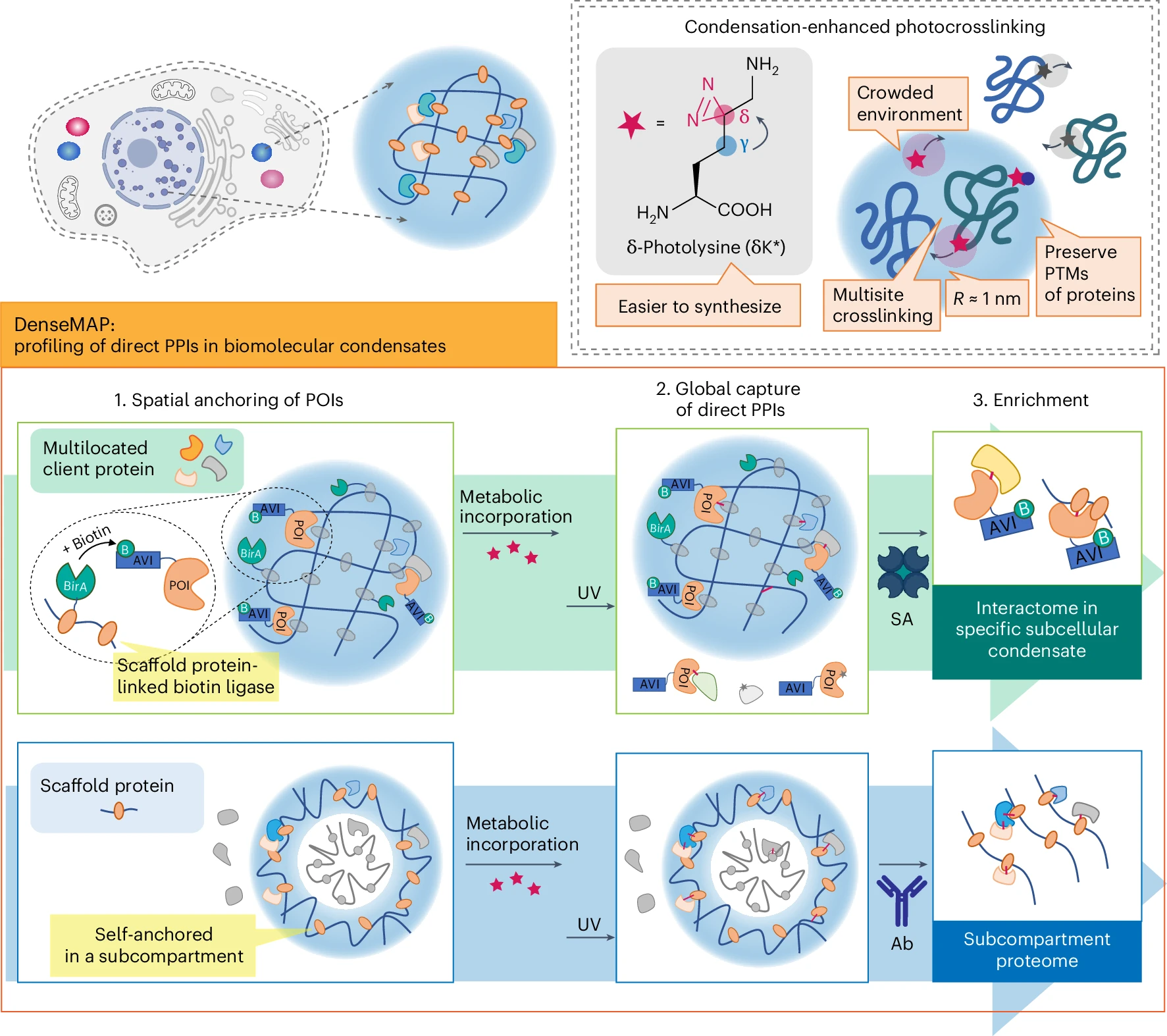
2.(Nat Chem,IF:19.2)通过凝聚增强光交联进行时空蛋白质相互作用组分析

11月5日,北京大学化学与分子工程学院陈鹏团队、北京大学基础医学院李婷婷团队,在 Nature Chemistry 合作发表研究,提出了一种通过凝聚增强光交联的时空蛋白质相互作用组分析新方法DenseMAP。
DenseMAP通过改进的光交联探针,实现了对生物分子凝聚体内部蛋白相互作用的时空分辨分析。DenseMAP结合δ-光赖氨酸探针和BirA蛋白标记,能够在1纳米范围内高效捕捉凝聚体中多位点的直接蛋白相互作用。
实验中,DenseMAP成功绘制了SARS-CoV-2 N蛋白在应激颗粒(SG)中的相互作用网络,揭示了磷酸化在病毒复制调控中的作用;同时,在核仁蛋白NPM1的动态互作中,发现SUMO化在核仁蛋白质稳态中的关键性。相比于传统方法,DenseMAP提高了特定亚结构内的蛋白相互作用精确度,为研究无膜细胞结构中的功能性蛋白网络提供了新平台。
3.(Eur J Heart Fail,IF:16.9)蛋白质生物标志物预测心力衰竭进展的多队列研究

10月28日,瑞典隆德大学的研究团队在 European Journal of Heart Failure 发表了心衰(HF)的蛋白质组学研究。
文章研究了在三个流行病学队列中蛋白质生物标志物与无症状性左心室肥厚(LVH)、舒张功能障碍(DD)及心力衰竭(HF)发生的关系。在1500余名参与者的MPP队列(Malmö Preventive Project)中,NT-proBNP与LVH(OR=1.48)、DD(OR=1.71)及心力衰竭(HR=1.98)均显著相关,且在STANISLAS和HOMAGE队列中得到一致验证。
此外,ST2、GDF-15和Galectin-4与新发心力衰竭显著相关。路径富集分析揭示了炎症和病毒感染可能是心力衰竭的潜在进展途径。
研究表明,NT-proBNP作为无症状性心脏功能障碍和心力衰竭风险预测的关键生物标志物,联合其他蛋白质标志物可进一步提高预测准确性。
更多心衰蛋白质组研究:
晚年心衰和虚弱的分子基础
https://jamanetwork.com/journals/jamacardiology/article-abstract/2819212
心力衰竭患者接受SGLT2抑制治疗后的蛋白质组学特征
https://www.jacc.org/doi/10.1016/j.jacc.2024.07.013
大规模血浆蛋白质组学鉴定了与心力衰竭发展相关的新蛋白质和蛋白质网络
https://www.nature.com/articles/s41467-023-44680-3
4.(Angew Chem Int Ed Engl,IF: 16.1)TEAQ:通过DIA-MS发现并筛选临床级生物标志物

10月21日,洛杉矶雪松西奈医学中心的研究团队在 Angewandte Chemie International Edition 发表研究,探讨了通过质谱技术(尤其是DIA-MS)发现并筛选临床级生物标志物候选物的流程。
文章提出了TEAQ软件包,用于从数据非依赖性采集质谱(DIA-MS)数据中筛选符合临床级别的生物标志物候选物。TEAQ通过线性、特异性、重复性等标准筛选DIA-MS数据中的前体、肽段和蛋白质,并应用于492例炎症性肠病患者的数据分析,识别出了1179种蛋白中的326种可量化蛋白质(1110个特征肽段)。TEAQ显著提高了生物标志物开发的准确性和效率,有助于加速临床验证与应用。
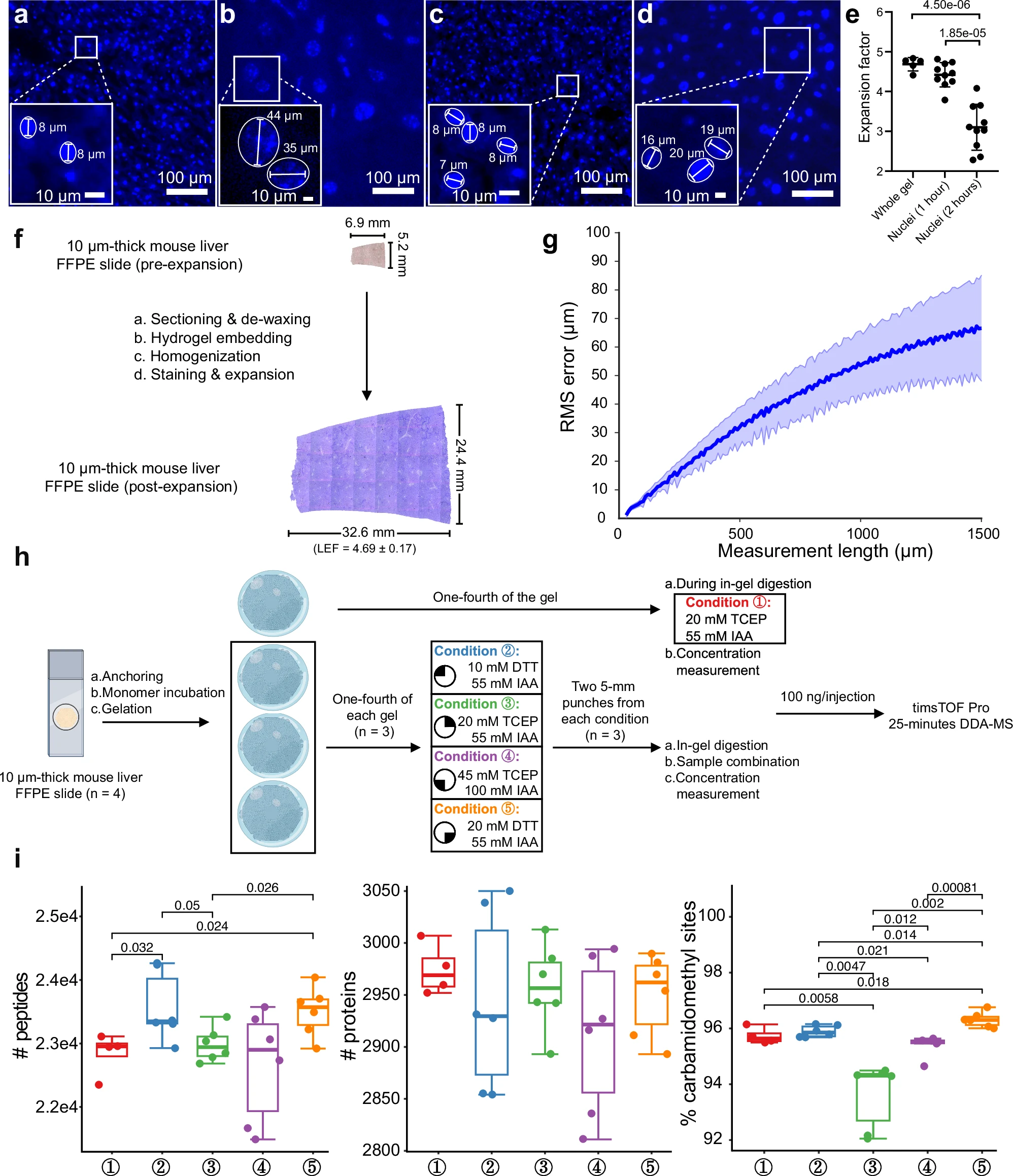
5.(Nat Comm,IF: 14.7)FAXP:石蜡切片中单个亚细胞器的深度蛋白质组分析

10月30日,西湖大学郭天南、Kiryl D. Piatkevich 团队合作在 Nature Communications 上发表研究,介绍了滤膜辅助膨胀蛋白组学(FAXP,Filter-aided expansion proteomics)技术在空间蛋白组学中的应用。
FAXP是西湖大学科研团队在空间蛋白组学领域的 “进阶版” 成果。FAXP在2022年研究的原有基础上进行了一系列步骤优化(包括切片制备、组织孵育、组织变性与染色等),以加速样品处理的时间、提高样品制备的通量、提升处理样本分辨率,并使其与临床FFPE组织样本相兼容,首次实现了石蜡切片中单个细胞核形状的深度蛋白质组分析。
FAXP方法结合了基于水凝胶的组织膨胀、滤膜辅助的样本制备流程和基于质谱的蛋白质组学,使得研究者可以在组织水平进行高空间分辨率的蛋白质表达分析,为深入理解组织中蛋白质的表达提供了有力工具,并在结直肠癌(CRC)研究中展示了其应用潜力。
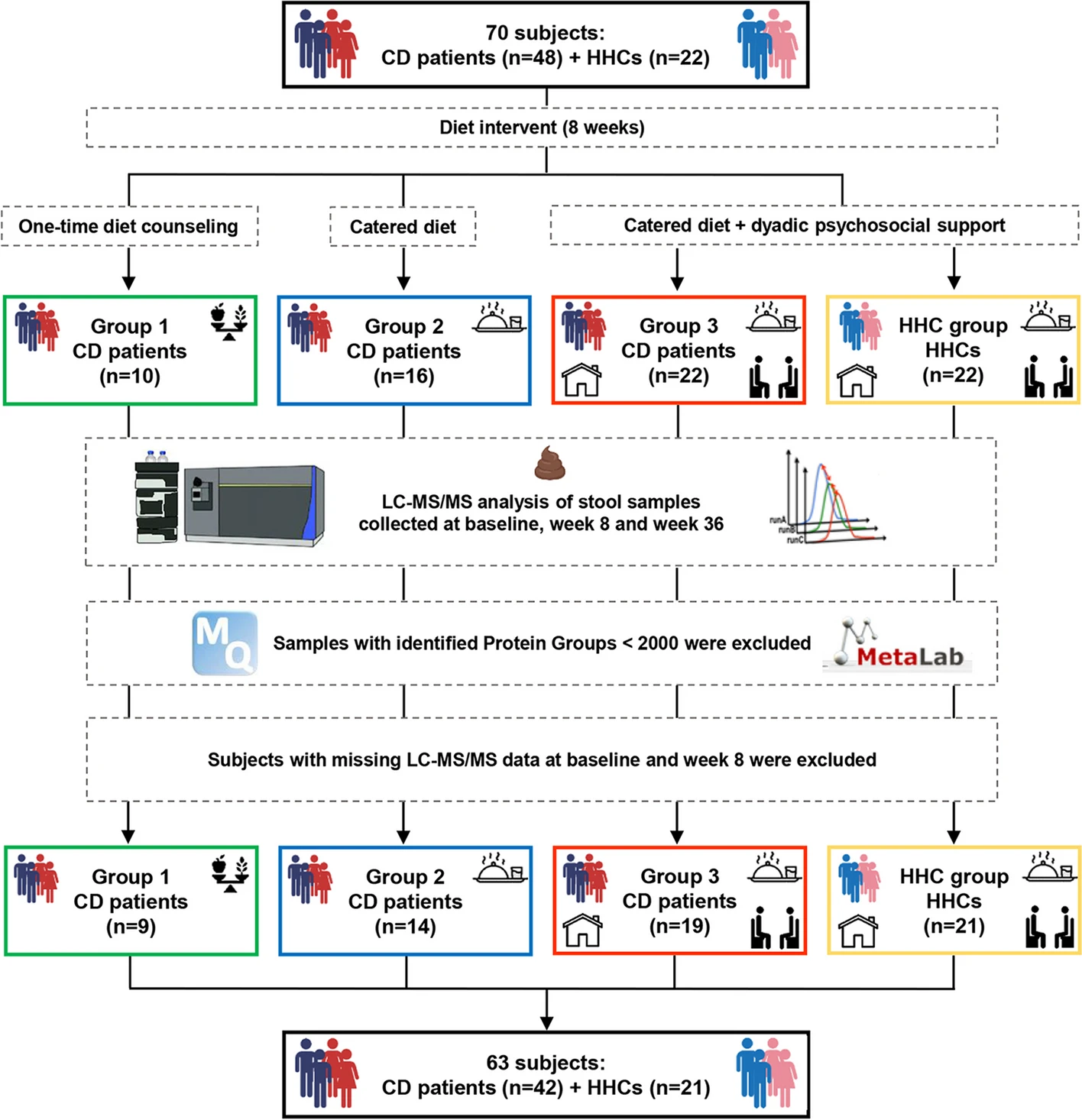
6.(Microbiome,IF: 13.8)宏蛋白组学揭示克罗恩病患者饮食干预的肠道微生物组功能变化

10月23日,罗马 Bambino Gesù 儿童医院和迈阿密大学米勒医学院的研究团队在 Microbiome 发表了克罗恩病(CD)的宏蛋白组学研究。
文章通过宏蛋白组学分析探讨了高纤维低脂饮食(Mi-IBD饮食)对克罗恩病患者肠道微生物群(GM)功能的影响。研究团队收集了来自70名参与者(48名CD患者和22名健康家庭对照)的209份粪便样本,样本分别来自基线、第8周(定制饮食结束)和第36周(恢复自由饮食)三个时间点,并通过液相色谱-串联质谱(LC-MS/MS)进行宏蛋白组学分析。
研究发现,在8周干预后,Mi-IBD饮食显著增强了CD患者肠道中碳水化合物代谢和铁的运输功能,但CD患者的碳水合成及嘌呤/嘧啶合成通路相较于健康对照(HHCs)仍显著缺乏。进一步分析表明,不同CD类型的微生物群响应不同:结肠型患者获益较大,而回肠型患者中抗炎菌种Faecalibacterium的丰度减少。
研究证明,饮食干预能有效改善CD患者的肠道微生物功能,但具体效果与疾病部位有关,提示未来的饮食干预应针对CD不同亚型进行分层研究。
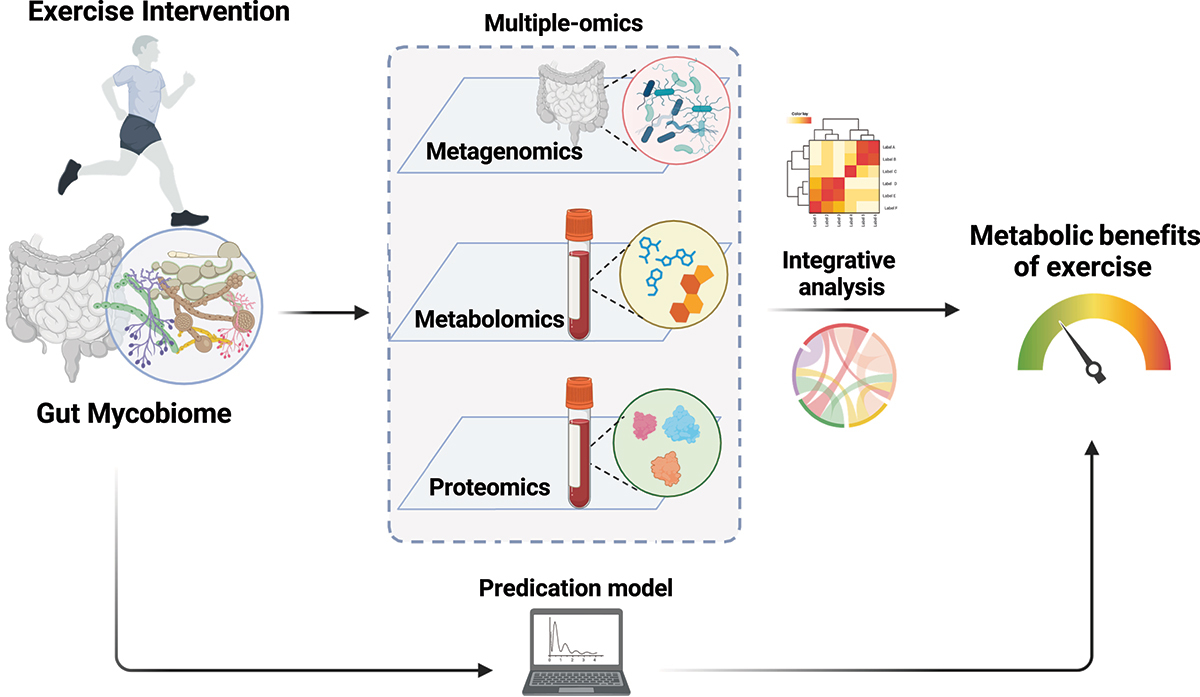
7.(Gut Microbes,IF: 12.2)多组学研究揭示运动改变肠道真菌群促进代谢健康

10月29日,香港大学徐爱民团队与德国汉斯诺尔研究所的研究团队一起,在 Gut Microbes 发表了一项多组学研究,探讨运动如何通过改变肠道真菌群(mycobiome)来产生代谢益处,从而帮助预防糖尿病。
研究基于39名中国男性的随机对照试验,结果显示,经过12周的运动干预,肠道真菌的多样性显著增加,且出现了如Verticillium等具潜在代谢益处的真菌属。
多组学分析显示,肠道真菌群的变化与代谢表型、细菌微生物组、循环代谢及蛋白组学特征之间存在复杂关联。同时,基于基线微生物特征构建的机器学习模型能有效预测胰岛素敏感性改善的运动响应性,训练组与验证组的ROC曲线下面积(AUROC)分别为0.91和0.79。研究表明,肠道真菌群或为运动代谢效益的关键传导因子,为个性化糖尿病预防干预提供了新的方向。
8.(Cell Genom,IF:11.1)AI赋能的微扰蛋白质组学:探索复杂生物系统的PMMP流程

11月1日,西湖大学郭天南团队,联合哈佛医学院 Chris Sander、苏黎世联邦理工学院(ETH Zürich)Peter Bühlmann、Ruedi Aebersold ,在 Cell Genomics 发表了一篇 Perspective 文章。
文章围绕AI赋能的微扰蛋白质组学(AI-empowered perturbation proteomics)在复杂生物系统研究中的前景,提出了一种全新的PMMP研究流程(微扰-测量-建模-预测),以应对当前系统生物学领域面临的数据不足和建模局限。
通过引入多种生物、化学和物理层面的微扰手段,并结合高通量蛋白质组学、转录组学和代谢组学等多模态数据,研究者能够更加精准地预测生物系统对微扰的响应。利用AI模型,微扰蛋白质组学可以鉴定药物机制、探索未充分研究蛋白质的功能、预测扰动响应,比如单药敏感性及双药协同作用等,进而协助设计个性化治疗方案。









