研究背景
在临床医学中,预后指的是具有特定疾病或健康状况的人未来健康结局的风险。因此,预后研究是对具有给定基线状态的人群进行观察,从而挖掘基线特征与未来结局之间的关系,比如通过对患者一些临床指标的检测,来预测其术后生存期的长短,或者是否对某种药物治疗产生响应。预后研究结果为临床决策、发现和评估患者治疗的新方法提供了相关证据。
随着质谱等技术的不断发展,蛋白质组学技术被越来越多地应用到预后研究当中,例如2019年[1],Nature 报道了一篇关于首次基于蛋白质组图谱对早期肝细胞癌进行精准分子分型的文章,研究发现了一批新的肝细胞癌早期诊断、预后评估和精准分型的生物标志物和药物治疗靶点,这些从蛋白质组变化中鉴定的药靶蛋白不仅被证明具有强大的识别不良预后肝癌患者的作用,而且还会使这些患者得到进一步的靶向治疗。
今天我们分享2篇基于蛋白质组学探索肿瘤预后评估标志物的科研论文,希望能为各位研究人员带来新的科研思路和灵感。
案例分享
案例1:
Cancer Letters | PulseDIA蛋白组发现直肠癌新辅助放化疗预后标志物[2]
文章概述:
2023年5月,浙江大学医学院第二附属医院肿瘤研究所和山东第一医科大学山东省肿瘤医院肿瘤研究所共同在 Cancer Letters 上发表了题为 “FOSL2 promotes intertumoral infiltration of T cells and increases pathological complete response rates in locally advanced rectal cancer patients” 的研究论文。
在这项研究中,研究人员纳入了浙江大学医学院第二附属医院(SAHZU)和山东省肿瘤医院肿瘤研究所(SCHI)两个中心,共计58名接受新辅助放化疗(nCRT)的局部晚期直肠癌(LARC)患者,通过压力循环技术(PCT)和脉冲式数据非依赖采集技术(PulseDIA)对患者活检组织FFPE样本进行蛋白质组学分析。
综合两个中心的数据和MRM靶向蛋白质组学验证,以及另一组54名LARC患者nCRT前活检队列的免疫组织化学(IHC)结果,发现了FOSL2能够反应nCRT的治疗效果,可以作为nCRT功效的新型预后生物标志物。并通过一系列机制研究,证实了FOSL2可以增强对nCRT的敏感性,并增加CD8+T细胞的浸润和杀伤以限制其他实体瘤的生长。西湖欧米为该研究提供了PulseDIA蛋白组、靶向蛋白组技术支持。

图1 论文截图
研究样本
(1)研究队列和样本:来自SAHZU和SCHI两个中心,58名接受nCRT治疗前LARC患者的88例FFPE样本(其中30例生物重复用于质控)进行发现蛋白组,54例样本进行IHC验证。
(2)组学方法:PulseDIA蛋白质组,MRM靶向蛋白组和免疫组织化学染色。
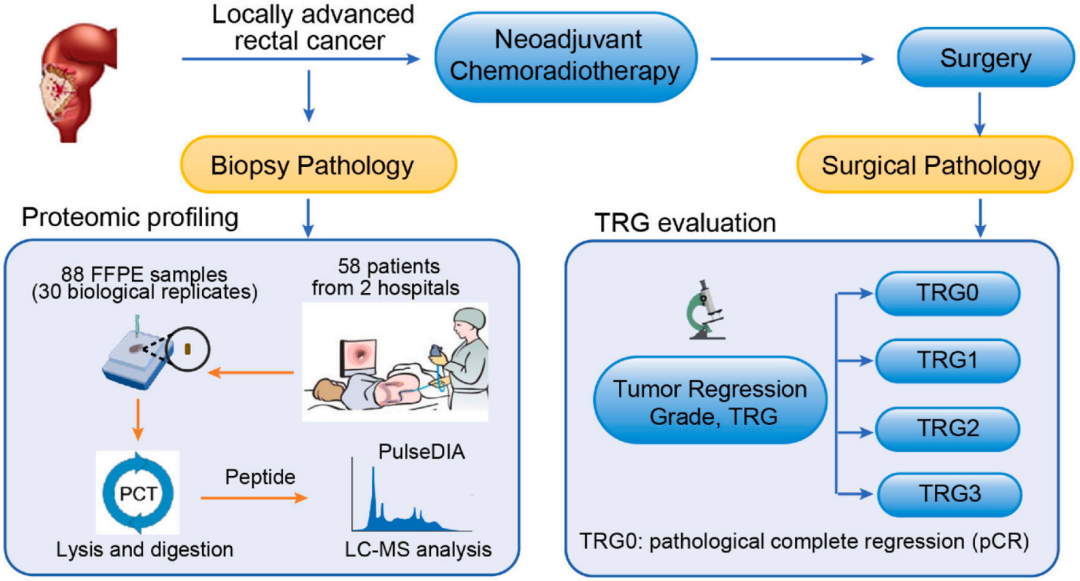
图2 技术路线图
相关研究结果
研究人员结合高效的PCT辅助样品制备方法和PulseDIA技术(西湖欧米独家专利)来实现在极少量的样本中鉴定更多的蛋白质,每个患者仅用约0.5–0.8mg的FFPE活检组织,便定量到6483个高可信度蛋白。两个中心之间共有3个上调蛋白(FOSL2、VSNL1、JUNB)和22个下调蛋白存在重叠(图3 A),并且FOSL2在肿瘤消退等级(TRG)为完全缓解(pCR)的患者中显著高表达(图3 B)。
MRM靶向质谱验证了PulseDIA中FOSL2在pCR组中高表达的结果(图3 C),并且FOSL2的MRM定量比PulseDIA定量具有更好的pCR预测能力(AUC:0.939与0.905,图3 D)。为了进一步研究FOSL2作为预测pCR的预后生物标志物的潜力,研究人员通过对另一组54名LARC患者进行IHC染色,验证了pCR患者中FOSL2的表达高于非pCR患者(图3 E)。
此外,通过MRM检测到FOSL2高表达的患者具有更长的无病生存期(DFS,FOSL2高与FOSL2低的中位DFS:1296天和784天,p=0.044,图3 F)。总的来说,这些结果表明FOSL2表达可用于预测pCR,作为nCRT治疗预后的标志物。
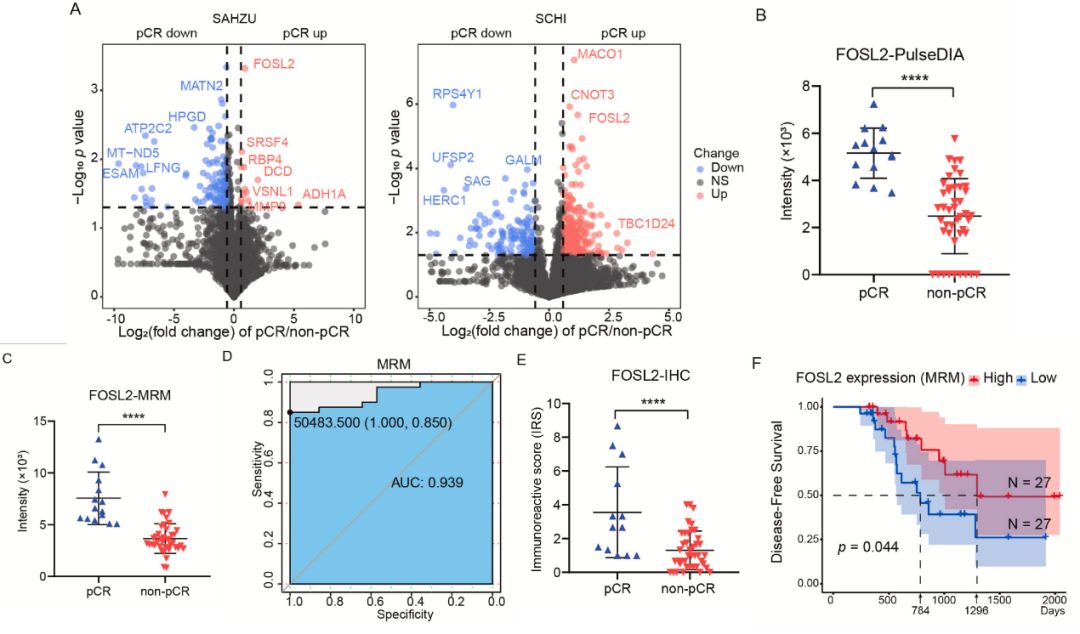
图3 FOSL2高表达可预测nCRT治疗效果
后续的功能实验证明,FOSL2可能会通过受辐射肿瘤细胞中胞质dsDNA的积累来触发CXCL10分泌,以招募更多CD8+ T细胞,产生颗粒酶B和IFN-γ,进而促进nCRT诱导的抗肿瘤免疫(图4)。
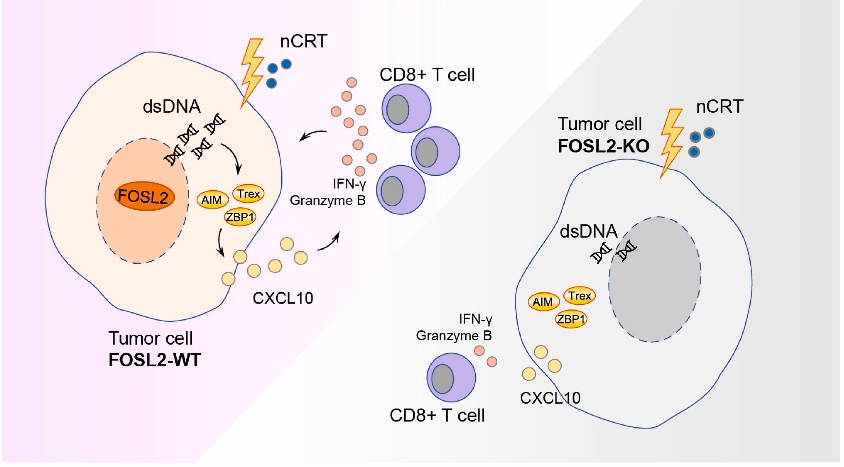
图4 FOSL2通过触发CXCL10分泌提升抗肿瘤效果
案例分享
案例2:
Nature Communications | 空间蛋白组学探索胶质母细胞瘤预后标志物[3]
文章概述:
2022年11月,法国里尔大学等研究团队在 Nature Communications 上发布了一篇名为 “Spatial analysis of the glioblastoma proteome reveals specific molecular signatures and markers of survival” 的文章,研究团队通过运用MALDI-MS和Label-free的空间蛋白组学分析了96名不同生存期胶质母细胞瘤(GBM)患者,确定了5种预后蛋白标志物,这5种蛋白质的表达是短期和长期生存的指标,可以帮助对患者进行分层。
研究结果强调了空间分辨蛋白质组学在破译GBM分子异质性和鉴定与生存相关的标志物方面的潜力。

图5 论文截图
研究样本
实验样本:46例胶质母细胞瘤样本
空间区域分割:红色区域,黄色区域,蓝色区域
HE染色后病理感兴趣的区域:
肿瘤区、非常致密和密集的浸润区、坏死区和微血管增殖(MVP)区
实验方法:MALDI-MSI, Label-free空间蛋白质组学
相关研究结果
考虑到GBM的异质性,研究者在质谱成像(MSI)的指导下对来自前瞻性患者队列的46个肿瘤进行了空间分辨蛋白质组学研究。
为了解患者在队列中的分层情况,研究者考虑了全局分割,考虑主要分子差异的分割图,确定了3个主要区域,红色区域A、黄色区域B和蓝色区域C。每个彩色区域都具有共同的分子特征,说明这些区域的光谱是相似的(图 10)。
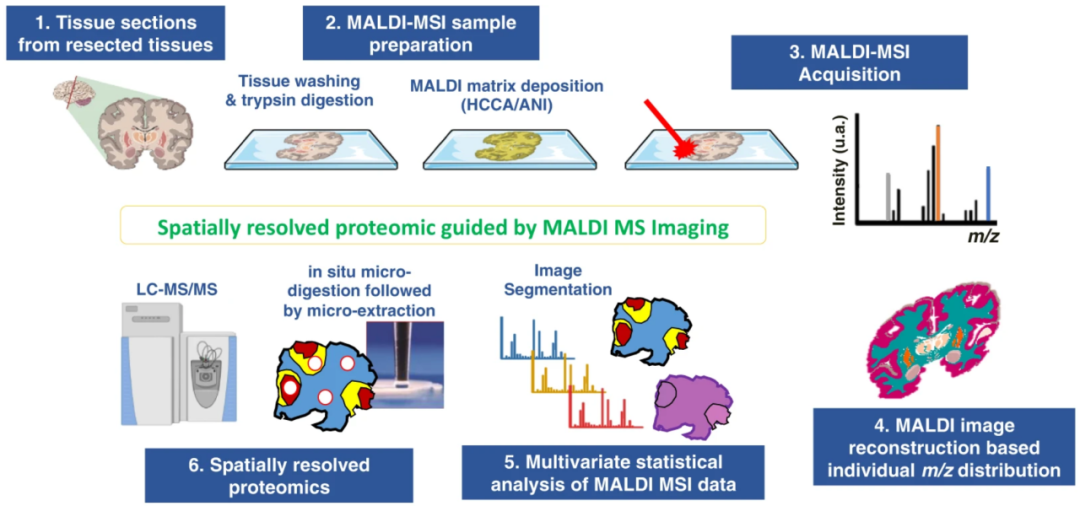
图6 技术路线图
使用label-free对这些提取样本进行蛋白质组学分析,共鉴定出4936种蛋白质。研究者首先通过皮尔逊相关分析测定了46个GBM样本中所有提取点之间的相关性,通过此项分析,确认了不同的蛋白质组学GBM亚型的存在,交叉验证了MALDI-MSI的结果。进一步发现,共有1183种蛋白质在三组之间显著差异表达(图11)。
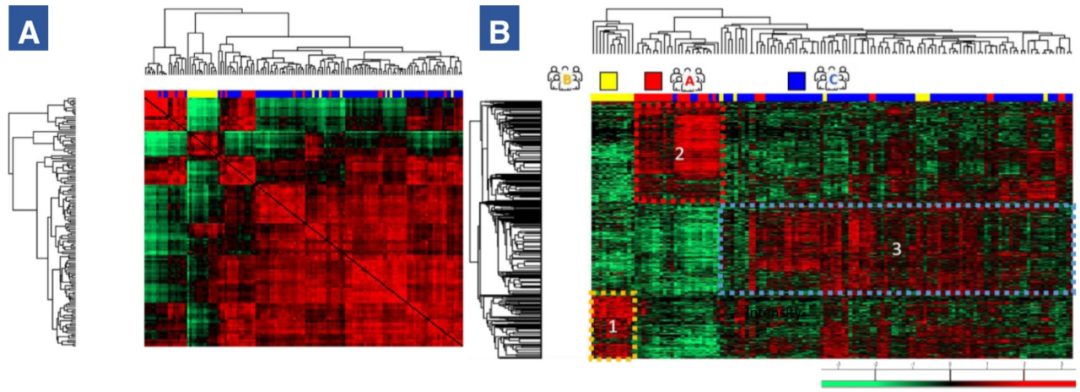
图7 空间分辨率的Label-free蛋白质组学
为了寻找潜在的预后蛋白,研究人员根据总生存期(OS)对整个蛋白质组数据集进行ANOVA检验,根据患者的OS将队列分为三组,采用Cox模型,发现5种蛋白质与患者生存期保持高度显著相关性(图12 A)。基于这5种蛋白质的表达,识别出2类患者(簇1和簇2)且OS差异显著(图12 B&C)。
在5种与生存期高度相关的蛋白质中,IP_652563、ALCAM和ANXA11是肿瘤的不良预后标志物,而PPP1R12A和RPS14是良好预后的标志物。
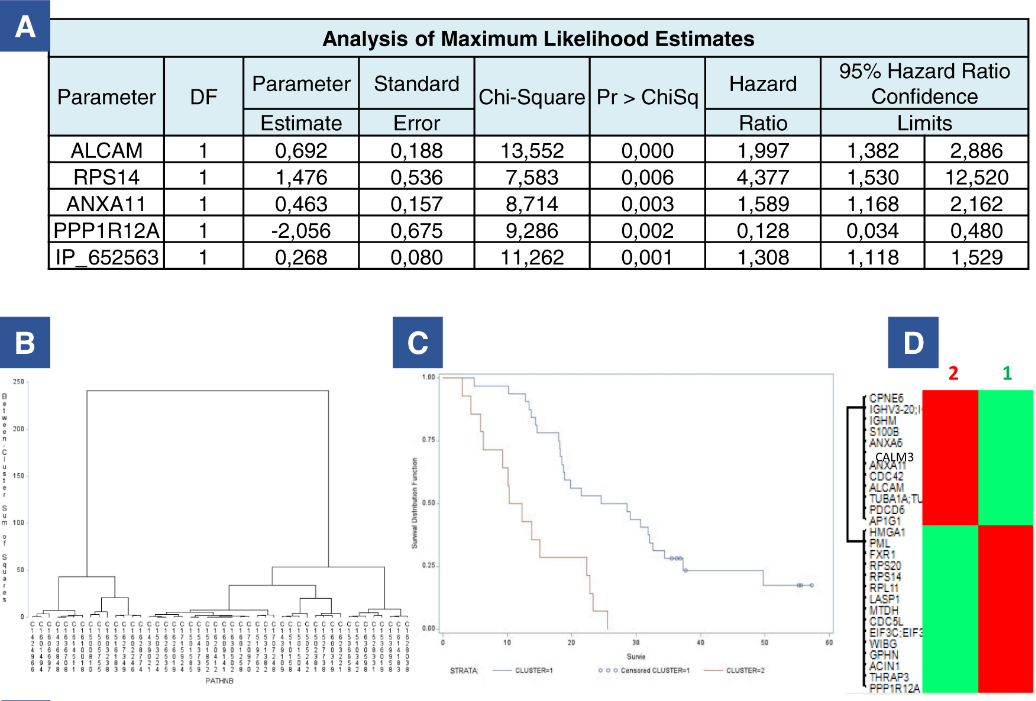
图8 模型性能测试和验证
研究者进一步通过免疫组织化学对50名患者的独立回顾性队列进行5种蛋白质的评估。根据患者的生存时间进行分组,分为三组不同的生存期。13例患者生存期较低(少于1年),25例患者生存期中等(1-2年),12例患者生存期高(2年以上)。
与低和中等生存期患者相比,RPS14和PPP1R12A在较长生存期患者的肿瘤中表达水平较高,与生存期较长的患者相比,ANXA11在低生存期和中等生存期患者的肿瘤中表达水平更高,证实了预后标志物的有效性。
西湖欧米预后分析研究套餐
西湖欧米专注于临床蛋白质组研究,包括高深度血液蛋白组学、空间蛋白质组学、微量蛋白质组学、宏蛋白质组学等特色业务。欧米核心技术是AI赋能临床样本的高通量蛋白质组分析技术,支持多种类型和极小临床样本量的蛋白质组学分析,可得到高重现性、低交叉污染的优质质谱数据。
截止目前,西湖欧米合作伙伴已在 Cell、Immunity、Cell Discovery、Molecular Cell、Cell Reports、Nature Commnuications、Cell Reports Medicine、Nature Protocols 、Molecular & Cellular Proteomics 等多类杂志上发表文章。
西湖欧米预后分析套餐,涵盖丰富的预后分析内容,包括机器学习筛选预后标志物、基于蛋白质组学数据的分子分型分析,以及从差异蛋白中筛选与患者生存相关的候选基因,进行多因素COX模型分析,ROC曲线评估风险模型的预测能力,KM评估模型的风险评分是否有意义等等。
感兴趣的老师赶紧联系我们吧。

参考文献:
[1] Jiang Y, Sun A, et al. Proteomics identifies new therapeutic targets of early-stage hepatocellular carcinoma[J]. Nature, Springer Science and Business Media LLC, 2019, 567(7747): 257–261.
[2] Xu K, Yin X, Zhou B, et al. FOSL2 promotes intertumoral infiltration of T cells and increases pathological complete response rates in locally advanced rectal cancer patients[J]. Cancer Letters, Elsevier BV, 2023, 562: 216145.
[3] Duhamel M, Drelich L, Wisztorski M, et al. Spatial analysis of the glioblastoma proteome reveals specific molecular signatures and markers of survival[J]. Nature Communications, Springer Science and Business Media LLC, 2022, 13(1).