蛋白质通常以大分子组装体的形式存在,这些组装体对调控和催化大多数细胞过程至关重要。然而,传统的亲和纯化结合液相色谱-串联质谱(AP–MS)方法不能在单个实验中解决复杂的异构体组成问题,需要从多个时间和资源密集型的反复AP–MS实验中进行计算推断。
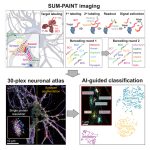
为了解决这一问题,苏黎世联邦理工学院分子系统生物学研究所的 Matthias Gstaiger 团队引入了一种称为 “深度交互分析质谱(deep interactome profiling by mass spectrometry,DIP-MS)” 的新方法,结合了AP和BN-PAGE*分离(blue-native-PAGE separation)、数据非依赖性采集和深度学习信号处理。
* BN-PAGE:Blue Native PAGE,Hermann Schägger 等人于1991年建立的蛋白电泳技术,用于从生物样品(质膜、胞浆等)中分离蛋白质复合物。
3月26日,该研究以DIP-MS: ultra-deep interaction proteomics for the deconvolution of protein complexes为题发表在Nature Methods(IF:48)上。

图1 论文截图
研究人员将DIP-MS用于对蛋白质复合物进行深入的相互作用蛋白质组学分析,以解析复合物的组成。文章通过质谱技术和结构预测揭示了细胞中新的蛋白折叠底物与PAQosome和PFD(prefoldin)复合物的相互作用网络,拓展了对细胞蛋白质折叠调控机制的认识。
DIP-MS方法流程概述
首先,通过亲和纯化将含有诱饵蛋白(bait protein)的蛋白质复合物富集,然后使用BN-PAGE将复合物按照表观分子量进行分离。
接下来,将凝胶切割成大约70个1mm宽的片段,然后使用96孔板格式的快速、可重复的filter aided in-gel消化制备方案对每个片段进行处理。
最后,使用短液相色谱梯度结合定量DIA-MS测量每个分数的蛋白质肽段。通过PPIprophet处理肽片段离子谱的共迁移矩阵来推断定量蛋白电泳洗脱模式、组装状态和PPIs。对显示多个峰的蛋白质轮廓进行进一步解卷积,从而在单个DIP-MS实验中鉴定包含诱饵蛋白的多个复合物。

图2 DIP-MS工作流程
结果1:
用于PPI预测和复杂推理的深度学习框架
PPIprophet是基于数据驱动神经网络的蛋白质复合物解卷积系统,可从DIP-MS数据中提取PPI、诱饵蛋白复合物的识别、亚单位数量和分子量的推断以及通常在AP-MS中看不到的prey–prey interactions。
研究人员开发了一个深度神经网络(DNN)模型用于PPI预测,并使用超过150万个从不同类型的共迁移测量中提取的PPIs进行训练。研究人员比较了PPIprophet与其他共迁工具的性能,并证明了其在DIP-MS数据集分析中的优越性。
结果2:
DIP-MS与AP-MS和SEC-MS工作流程的基准测试
研究人员比较了使用PFDN2作为诱饵蛋白的DIP-MS结果与两种正交方法生成的数据:SEC-MS和使用11个PFD-PFDL亚基作为诱饵的反向AP-MS数据集。
结果表明,DIP-MS数据共鉴定了353个相互作用伙伴,比SEC-MS和AP-MS多出187个,覆盖了大约30%的公共数据库中的相互作用伙伴。
研究人员进一步分析了DIP-MS与SEC-MS或AP-MS数据的拓扑结构和网络连通性,并发现DIP-MS生成的网络更加广泛、密集,并且重现了更多的真实情况。
结果3:
Prefoldin和prefoldin-like复合物的全局结构
研究人员使用DIP-MS分析了PFDN2和PFDL(prefoldin-like)中的UXT,并得到了详细的prefoldin和prefoldin-like复合物谱图。在两个DIP-MS实验中,研究人员总共分析了1513个蛋白质,检测到了6762(PFDN2)和5682(UXT)个高置信度二元相互作用,得到了包含11552个唯一相互作用和939个蛋白质的网络。
在组合DIP-MS衍生网络中,研究人员识别了所有已报告的PFD–PFDL组装,包括PFD、PFDL和包含PAQosome的PFDL。此外,他们还识别了由R2TP蛋白RUVBL1/2、RPAP3和PIH1D1以及CCT/TRiC-PFD复合物的所有亚基组成的共迁移组。

图3 从PFDN2和UXT DIP-MS实验中鉴定组装
结果4:
发现含PDRG1的另一种PFD复合物
研究人员发现了一个新的PFD复合物亚型,命名为PFD homolog(PFDh),其中包含了PDRG1。
通过DIP-MS实验和结构预测,研究人员确定了PDRG1与经典PFD亚基形成的复合物。进一步的验证表明,PDRG1与经典PFD亚基发生交互作用,并构成了PFD复合物的一个变体。结构预测显示PFDh与经典PFD有相似的结构,但PDRG1的存在可能影响其底物的特异性。该研究的发现揭示了PFD复合物的多样性,未来需要进一步研究PFDh的分子功能。
结果5:
PAQosome和PFDL核心成分的鉴定
通过DIP-MS和AP-MS方法,研究人员确定了PAQosome和PFDL的核心组分。他们发现了ASDURF作为PFDL的亚基,同时也是PAQosome超级复合物的成分之一。
此外,研究还揭示了POLR2E与PFDL复合物之间的PPI,表明POLR2E是PFDL复合物的核心组分之一,这与目前文献中关于PFDL复合物的六聚体范式(hexameric paradigm)存在偏差,并提出了一个更新的包含POLR2E的异质性七聚体PFDL复合物的假设。
结果6:
通过DIP-MS鉴定了经典PFD折叠客户
利用DIP-MS技术的高灵敏度,研究人员识别了与PFD和CCT/TRiC复合物相互作用的新型折叠客户(folding clients)。
通过从PPIprophet派生的PPI网络(10%FDR阈值)中选择与CCT/TRiC和PFD或PAQosome相互作用的蛋白质亚网络,识别了这些亚网络中的复合物-复合物相互作用以及蛋白质-复合物相互作用。
结果7:
鉴定PAQosome的客户复合物及客户
利用DIP-MS技术,研究人员鉴定了完全组装的PAQosome辅助伴侣的客户复合物(client complexes)和客户蛋白(client proteins)。PAQosome是一个大型多蛋白质组装体,协助HSP90组装蛋白复合物(如snRNPs、RNAP复合物和snoRNP组装体)。
研究人员发现了45个已知clients以及许多其他潜在clients,并通过共沉积和AP-MS数据验证了这些发现。此外,还发现了新的潜在客户复合物和客户蛋白,揭示了DIP-MS在解剖特定相互作用网络方面的分辨率和有效性。

图4 PAQosome客户及客户蛋白复合物的分配
总 结
总的来说,该研究介绍了一种新的蛋白质组学方法,为理解蛋白质复合物的组成和结构提供了一种高效且高分辨率的工具。
在文章讨论部分中,研究人员总结了DIP-MS的优点,并强调了其对于研究蛋白质组学和细胞蛋白质相互作用的重要性。他们还提出了一些可能的改进方向,以提高方法的准确性和适用性。
文章链接:
https://www.nature.com/articles/s41592-024-02211-y?utm_medium=external_display&utm_source=stork&utm_content=email&utm_term=null&utm_campaign=CONR_JRNLS_AWA1_CN_CNPL_0034V_STKRE