在生命科学研究中,单细胞蛋白质组学具有诸多优势,例如:能够鉴定耐药细胞中激活的途径,从而为癌症诊断和预后提供生物标志物。然而,其数据分析仍面临着肽段定量不确定性、数据缺失、批次效应和数据噪声等诸多问题。
3月19日,腾讯AI Lab和南开大学人工智能学院的研究人员在 Nature Methods(IF:48)合作发表了新的研究 scPROTEIN: a versatile deep graph contrastive learning framework for single-cell proteomics embedding,旨在解决单细胞蛋白质组学数据分析中的一系列挑战。
文章介绍了一种名为scPROTEIN的单细胞蛋白质组学数据分析框架,能够有效处理数据不确定性、批次效应和数据缺失问题,并在细胞聚类、数据整合、标签转移和空间分析等方面展现了优越性能。

图1 论文截图
scPROTEIN框架概述
scPROTEIN的整体框架包括三个阶段的工作流程。
第一阶段通过多任务异方差回归模型估计肽段信号的不确定性,将其聚合到蛋白质水平。第二阶段构建细胞图,利用图对单细胞蛋白质数据进行学习和降噪。第三阶段利用经过训练的图卷积网络编码器学习细胞嵌入,用于各种下游任务。
这一框架能够有效地处理肽段信号的不确定性、数据缺失、批次效应和数据噪声等问题。该方法在多种单细胞蛋白质组数据集上得到了验证,并展现了广泛的应用前景。
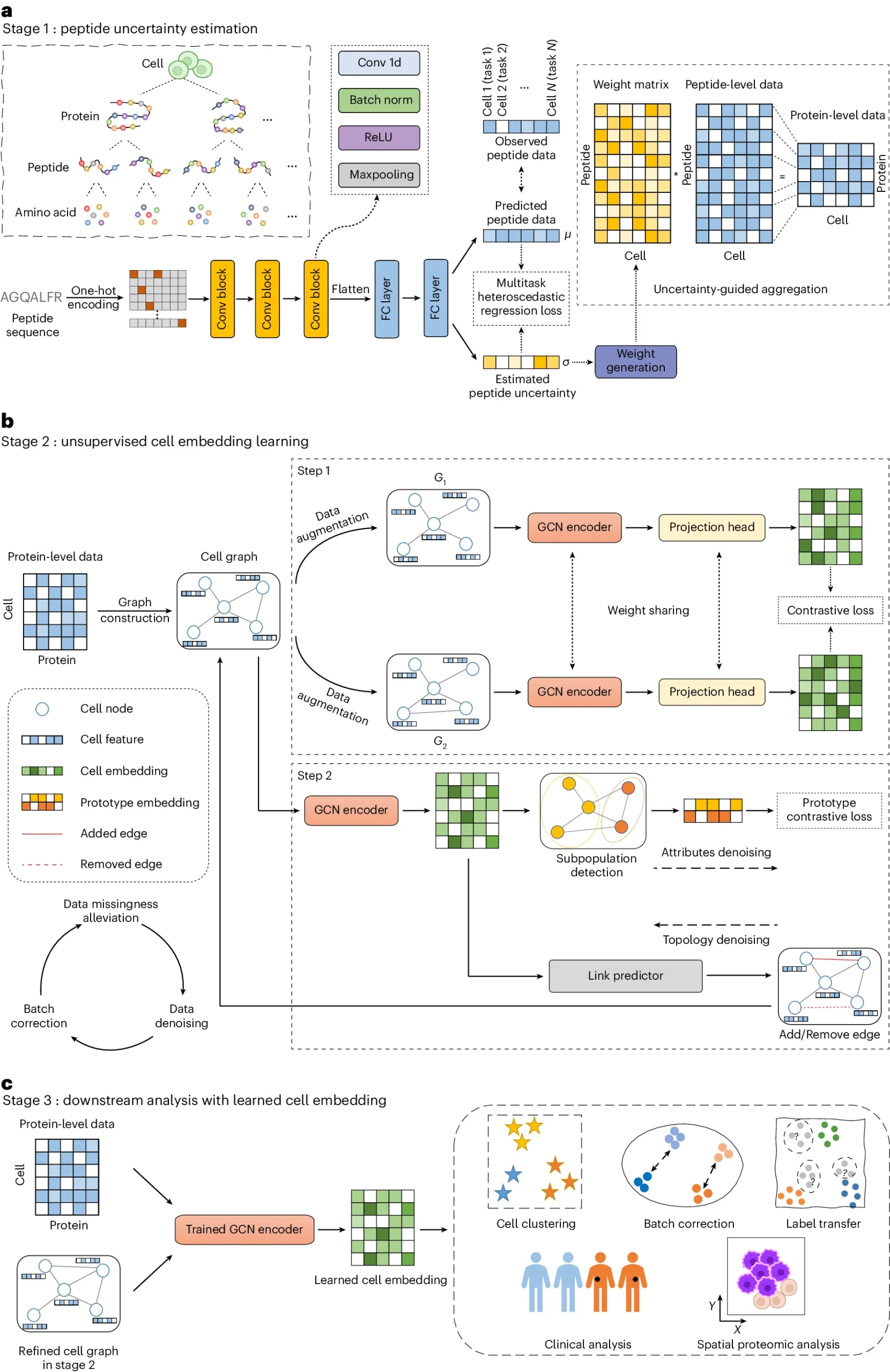
图2 a, 用于肽不确定性估计的多任务异方差回归模型;b, 基于深度对比学习的无监督细胞嵌入模型;c, 经过训练的图编码器使用推理来生成单元嵌入,然后将其应用于各种下游任务。
结果部分展示了scPROTEIN的性能表现以及在单细胞蛋白质组学数据分析中的应用情况,主要包括以下几个方面:
结果1:
细胞聚类和肽段不确定性估计
研究人员评估了由提出的方法学习的细胞嵌入在细胞聚类任务中的表现,并详细说明了scPROTEIN从第一阶段到第三阶段的工作过程。通过量化3,042种蛋白质的1,490个细胞的SCoPE2_Specht数据集上应用scPROTEIN,研究人员展示了整个学习流程,并与现有的单细胞蛋白质组学数据分析流程进行了比较。该方法在细胞聚类任务中表现出了最佳性能,并且相对于其他方法(MAGIC、AutoClass、Harmony、Scanorama和Liger),在评估指标上取得了更好的结果。
此外,研究人员还对蛋白质水平数据进行了分析,说明了如何通过scPROTEIN对蛋白质水平数据进行不确定性估计,以及不同批次和样本中的数据噪音情况。研究结果表明,scPROTEIN能够有效地估计肽段水平数据中的噪音,并生成更具信息量的蛋白质水平数据,进而提高了细胞嵌入的质量和下游任务的性能。
结果2:
实现数据整合和标签转移
scPROTEIN能够实现单细胞蛋白组数据的整合和标签转移(Label Transfer)。通过评估在不同单细胞蛋白组学平台引起的批次效应,研究人员发现,相比其他方法,scPROTEIN在去除批次效应方面更为稳健。实验结果表明,scPROTEIN在整合不同MS测序技术数据和标签转移方面具有良好的性能,能够准确地对细胞类型进行注释,展现了其在蛋白组学研究中的潜力。
结果3:
将scPROTEIN应用于临床蛋白质组学数据
研究人员将scPROTEIN应用于来自临床组织的单细胞蛋白质组学数据。这些数据来自ECCITE-seq数据集,涵盖了来自健康供体和T细胞淋巴瘤(CTCL)患者的6,500个细胞,量化了49个标记蛋白。
首先,研究人员集成了两个供体的数据以消除批次效应,并比较了scPROTEIN和其他竞争方法的批次校正性能。随后,通过细胞聚类和合并相似簇,他们将细胞分成了18个簇,并使用scPROTEIN学习的嵌入进行了分类。对三个代表性簇进行详细分析,并发现了CTCL细胞的生物标志物。其中,程序性死亡受体1(PD1)在研究模型的结果中明显上调,这表明在CTCL患者的细胞中检测到了PD1水平的增加。

图3 scPROTEIN 在临床蛋白质组数据集中的应用
结果4:
应用于空间蛋白质组学数据
研究人员还将scPROTEIN方法应用于BaselTMA数据集的单细胞空间蛋白质组数据分析中。通过构建细胞图和分析空间异质性,研究人员发现肿瘤样本显示出高度的区域化表型,而正常样本则表现出较高的细胞类型混合水平,这一发现与之前的乳腺肿瘤研究结果一致。研究证明,scPROTEIN可以有效地区分肿瘤样本和非肿瘤样本,并量化空间异质性。相比之下,仅使用原始蛋白质数据则无法实现此目标。
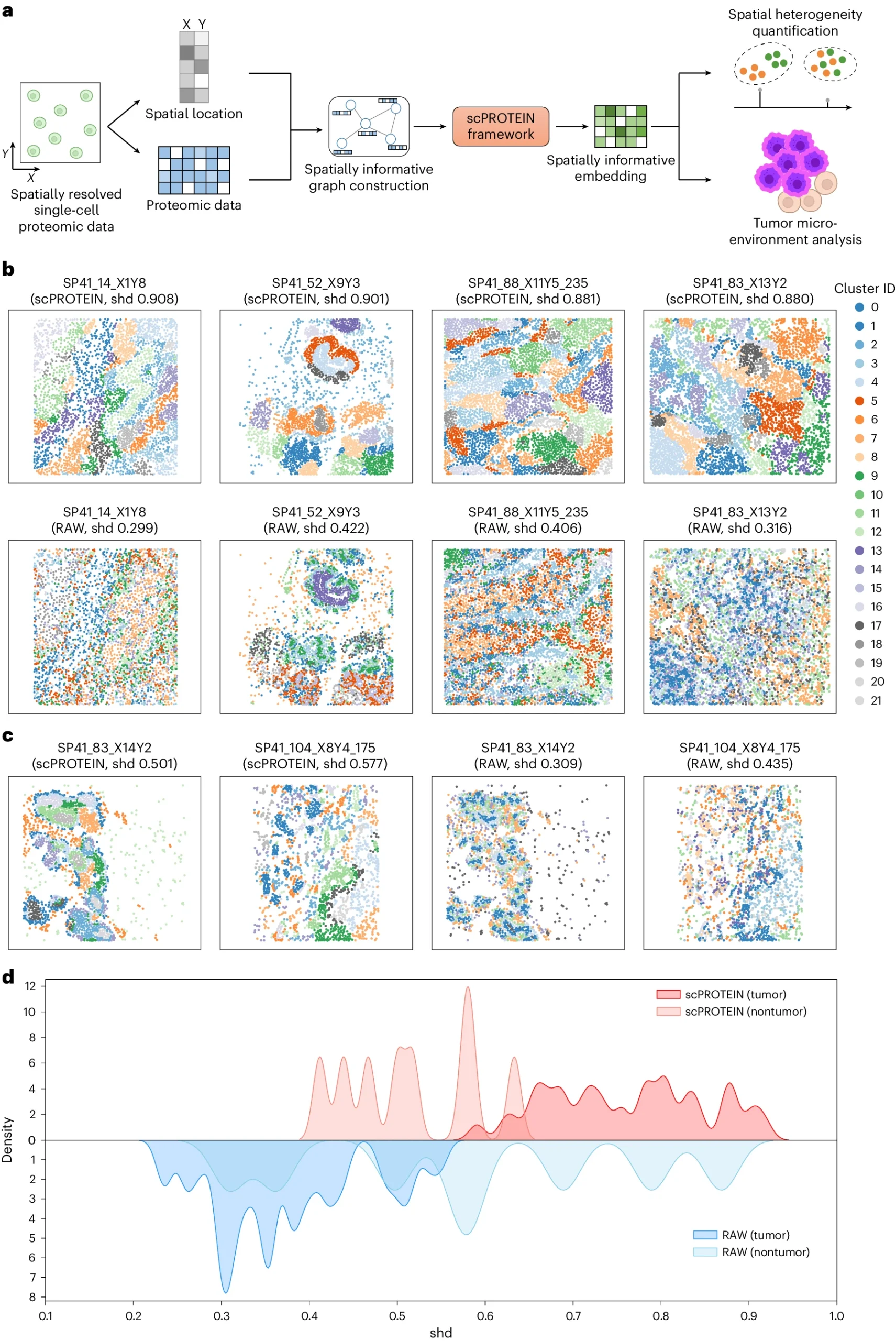
图4 scPROTEIN 在空间蛋白质组数据中的应用
总 结
总的来说,scPROTEIN在单细胞蛋白质组学数据分析中表现出了良好的性能和广泛的应用前景,有效地解决了单细胞蛋白质组学数据分析中的多个挑战,为深入理解细胞表型和疾病机制提供了有力工具。
该研究也存在一些局限性,例如部分质谱采集平台提供了直接从蛋白质水平提取的原始数据,而scPROTEIN工作流程的第一阶段无法适用于这种数据。
研究人员还展望了单细胞蛋白质组学数据处理领域的未来发展方向,包括更精确、直接、可信的基准数据集以及更多的单细胞蛋白质组学数据处理方法的发展。
文章链接:
https://www.nature.com/articles/s41592-024-02214-9?utm_medium=external_display&utm_source=stork&utm_content=email&utm_term=null&utm_campaign=CONR_JRNLS_AWA1_CN_CNPL_0034V_STKRE









