2025年3月25日,西湖大学医学院郭天南团队在 Cell Research 发表了题为 Grow AI Virtual Cells: Three Data Pillars and Closed-Loop Learning 的评述文章(editorial),探讨了人工智能虚拟细胞(AIVCs)的发展方向。
AIVCs的核心思想是通过人工智能和多模态数据整合,构建精确且可扩展的虚拟细胞模型。相比传统的虚拟细胞建模方法,AIVCs能够更全面地模拟细胞功能,并具有高通量仿真能力,甚至在某些情况下可以替代实验室实验。

图1 论文截图
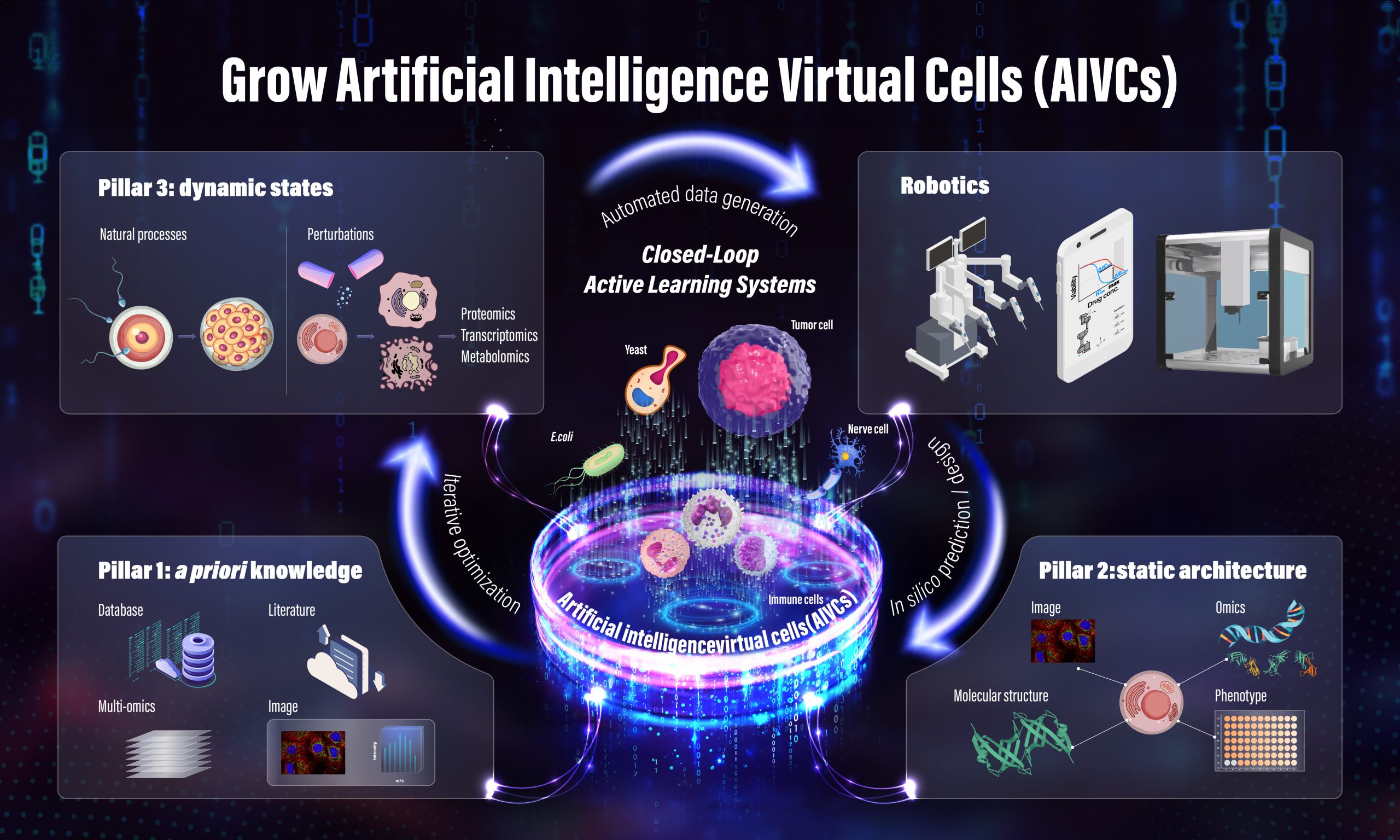
图2 AIVC构建框架 详见:guomics.com/way
提纲挈领
文章探讨了AI虚拟细胞(AIVCs)的构建方法与发展方向,提出了AIVCs的核心依赖于三大数据支柱——先验知识、静态结构和动态状态,并强调高通量组学数据(特别是微扰蛋白质组学数据)在动态模拟中的关键作用。
研究进一步提出了闭环主动学习系(Closed-Loop Active Learning Systems),结合AI预测与自动化实验,实现自适应优化,加速细胞建模与科学发现。为确保AIVC概念的可行性,研究人员建议从酵母(S. cerevisiae)等较简单但信息丰富的细胞模型入手,并逐步扩展到更复杂的人类癌细胞系,以推动AIVCs在生物医学、药物开发和个性化医疗中的广泛应用。
- 背景介绍 -
在生物医学研究中,细胞是生命的基本单位,对于理解健康、衰老、疾病以及药物开发和合成生物学至关重要。然而,传统的细胞实验通常需要消耗大量资源,并且实验结果易受变异影响,导致可重复性问题。因此,研究人员提出了虚拟细胞(Virtual Cells)或数字细胞(Digital Cells)的概念,以减少实验成本并提高研究的准确性和效率。
早期的虚拟细胞模型主要依赖于低通量的生化实验,并使用微分方程或随机模拟方法对特定的细胞过程进行建模。然而,这些方法在数据整合和动态模拟方面存在局限,难以全面描述细胞的复杂性。
随着高通量生物技术和人工智能(AI)的发展,人工智能虚拟细胞(AIVCs, AI Virtual Cells)成为一种新的研究方向,它结合了多模态数据和先进计算模型,为生物医学研究提供了新的可能性。
01
三大数据支柱:AIVCs的基础构建
为了更好地支持AIVCs的发展,研究提出了三大数据支柱(Three Data Pillars),作为AIVCs的核心数据基础:先验知识(a priori knowledge)、静态结构(static architecture)和动态状态(dynamic states)。这些数据结合AI算法,为虚拟细胞的构建提供必要的基础。
先验知识包括生物医学文献、分子表达数据和多尺度成像数据,涵盖细胞生物学的基本机制。尽管这些数据庞大且多样,但信息分散,难以直接用于构建完整的AIVC,因此只能作为基础框架。
静态结构是AIVC的第二个支柱,涉及细胞的形态学和分子组成,包括纳米尺度的分子建模、低温电子显微镜、空间组学等技术。这些数据能提供细胞的三维空间结构信息,但无法反映细胞的动态变化。
为构建真正 “活” 的AIVC,动态状态是必不可少的,它涵盖生理过程(如衰老、发育、疾病)以及外部微扰(如基因编辑、药物刺激)带来的影响。随着高通量组学技术(如转录组学、蛋白质组学、代谢组学)的发展,现在可以系统性地分析大量分子在不同细胞状态下的变化,提高AIVC的准确性。
文章指出,基于微扰的组学数据(perturbation-based omics data),包括转录组学(transcriptomics)、蛋白质组学(proteomics)和代谢组学(metabolomics),被认为是推动AIVCs发展的关键因素。而在这些数据中,微扰蛋白质组学数据尤为关键。
通过AI整合微扰数据,AIVC可以更精准地预测细胞如何对外部干预作出反应,为药物开发和细胞建模提供更强的支持。新兴的单细胞组学和空间组学技术也进一步增强了AIVC的动态模拟能力。
同时,文章指出,AIVC需要依赖AI驱动的多模态数据整合,结合深度学习技术(如变换器、CNNs)来解析复杂数据,最终推动系统生物学、个性化医学和药物研发的发展,为细胞行为研究提供新的视角
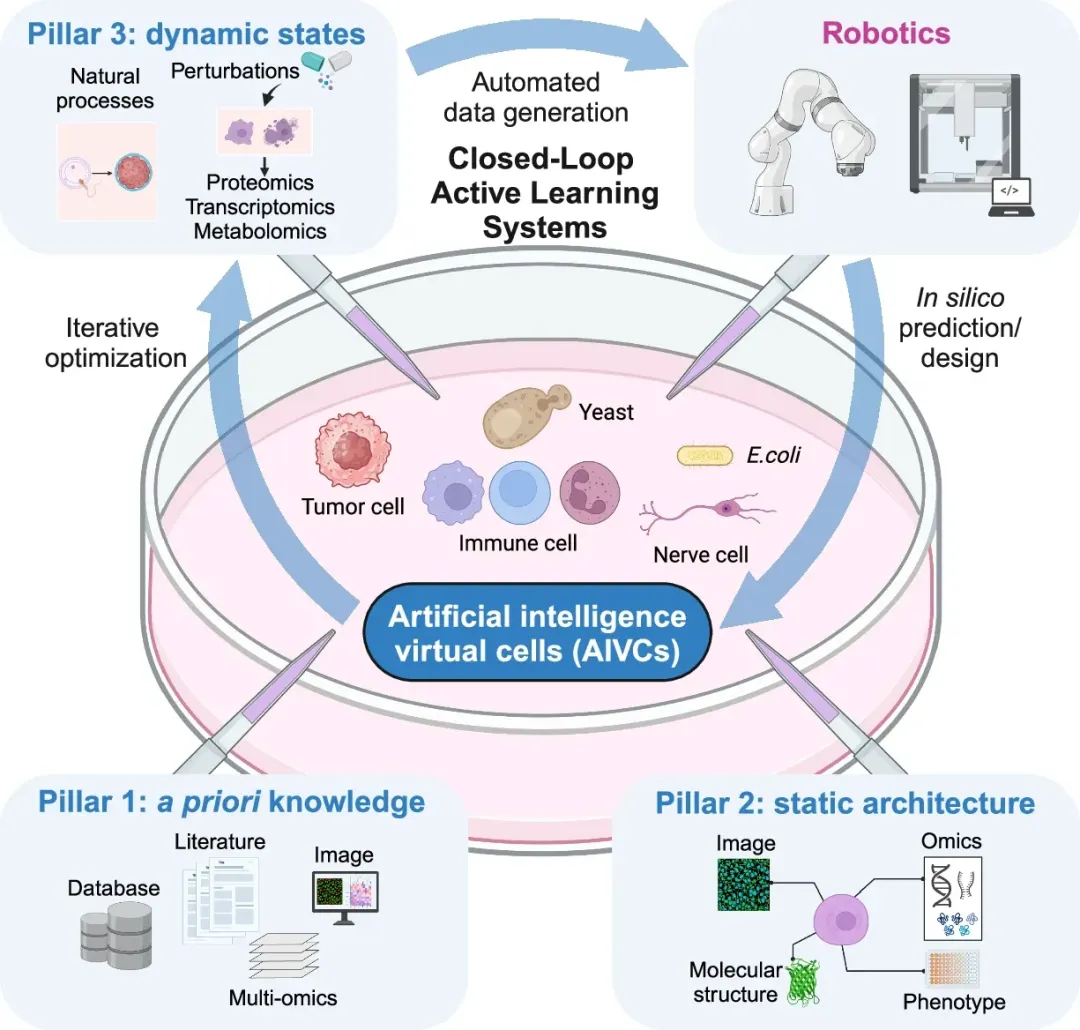
图3 通过闭环学习促进AIVC生长和发展的数据支柱
02
AIVCs的进化:闭环主动学习系统
AIVCs正在从静态、数据驱动的模型向自适应进化系统发展,其中闭环主动学习系统(Closed-Loop Active Learning Systems)是关键。
文章指出,传统方法依赖被动数据积累,而闭环系统结合AI预测与机器人实验,主动探索细胞动态状态,填补数据空白。这种系统能够自动识别知识缺口、设计实验、执行扰动,并实时优化模型,显著加速科学研究。
该系统的核心优势在于高效处理细胞对不同扰动的复杂响应。相比传统方法,AI可以优先选择最具影响力的实验(如CRISPR基因敲除、小分子药物处理等),最大化数据价值。例如,AI可能发现某些未知的磷酸化事件,并指导机器人实验以深入解析信号传导机制。
随着机器人实验和多模态数据整合的提升,AIVCs未来或可自主解析细胞生物学难题,如特定环境下的蛋白功能。这一方法标志着生物研究从被动观察向主动探索和自我优化的转变。
03
低门槛切入点:选择适合的细胞模型
AIVC的首个细胞模型选择至关重要,不同候选细胞各有优劣。文章指出,支原体(mycoplasma)较简化但通用性受限,大肠杆菌(Escherichia coli)数据丰富但缺乏真核复杂性,酵母(S. cerevisiae)兼具基因可操作性和真核特性,而人类癌细胞系在医学研究中数据广泛且与疾病相关。然而,这些模型在动态状态数据(尤其是扰动蛋白质组学)方面仍存在空白。
因此,研究人员建议从酵母入手——因为它既简单又包含真核细胞结构,数据相对丰富,并已在生物学和药物筛选领域广泛应用。研究人员把虚拟酵母细胞(Virtual Yeast Cell)作为AIVCs的入门方向,为后续研究奠定基础。
接着,人类癌细胞系则是后续重要目标,可推动AIVC在精准医学和药物开发中的应用。也就是说,先从简单模型入手,有助于优化AIVC的数据需求、建模策略和评估框架,为未来扩展到更复杂细胞系统奠定基础。
未来,AIVCs有望在药物开发、疾病建模和基础生物学研究中发挥重要作用,而科学界的协同合作对于推动这一领域的发展至关重要。因此,建立AIVCs的标准和最佳实践,将成为该领域下一阶段的重要任务,以确保AIVCs能够真正实现其在计算生物学和生物医学研究中的变革性潜力。
延伸阅读
1月12日,西湖大学郭天南团队,联合深势科技 / 北京科学智能研究院温翰团队,在bioRxiv在线发表了预印本文章 A perturbation proteomics-based foundation model for virtual cell construction。
研究建立了一个名为ProteinTalks的基础模型,通过整合大规模动态微扰蛋白质组数据和药物信息,可准确预测药物反应、协同效应和挖掘耐药分子机制,在精准医学领域具有广泛应用潜力。
具体来说,研究人员通过使用63种FDA批准的药物和其他抗癌化合物,针对18种乳腺癌细胞系进行药物处理(perturbation),产生了相应的蛋白质组学微扰数据集,并以此为基础构建了一个动态的蛋白质网络模型ProteinTalks。
这是第一次AI赋能的微扰蛋白质组学建立肿瘤细胞AIVC动态分子网络的试点研究。西湖大学医学院的研究人员表示:未来将探索建立更完善的AIVC,应用于药物研发、合成生物学、系统生物学等领域。








