生命科学研究的一个关键目标,是通过量化所有生物分子及其空间的相互作用来理解生物系统。
传统的蛋白质组学方法虽然在量化核酸和蛋白质丰度方面取得了显著进展,但要全面理解生物过程和功能,就需要空间组学技术,来提供绝对定量和亚细胞定位。
3月28日,马克斯·普朗克生物化学研究所的Ralf Jungmann团队及哥廷根大学医疗中心的Eugenio F. Fornasiero团队合作在Cell上发表了最新研究:Spatial proteomics in neurons at single-protein resolution。
图1 论文截图
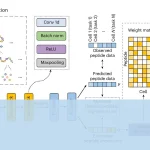
文章介绍了一种名为SUM-PAINT的新型成像技术,利用二级标记的DNA-PAINT实现了高度多重、单蛋白水平的超分辨率成像,为构建单蛋白水平的细胞图谱提供了新途径。
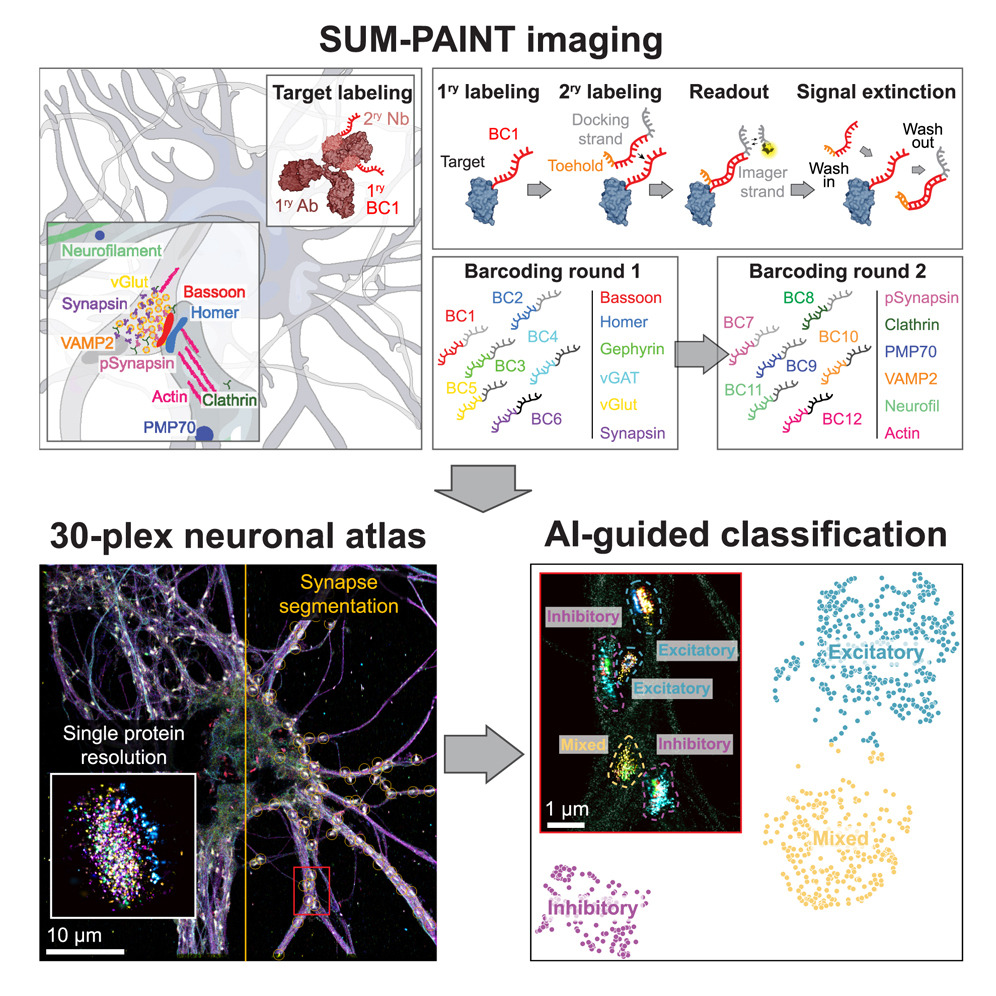
图2 图文摘要
文章结果部分主要介绍了使用SUM-PAINT技术在神经元中实现了高通量的单蛋白成像,并建立了一个30种蛋白质的单蛋白分辨率神经元图谱。
结果1:
基于二级标记的DNA-PAINT可实现单个蛋白的高度多重成像
研究人员采用了一种称为SUM-PAINT的方法,通过使用主条形码和二级标记(Secondary-label)将目标成像与多重化分离,提高了成像的多重性。此方法利用了速度优化的DNA-PAINT,以及具有二十个核苷酸序列的主条形码和与之互补的二级标记,实现了100倍的成像速度提升。
该方法可实现多达30个目标的成像,并具有较高的信号消光效率(extinction efficiency),为单蛋白水平的空间蛋白质组学提供了新途径。
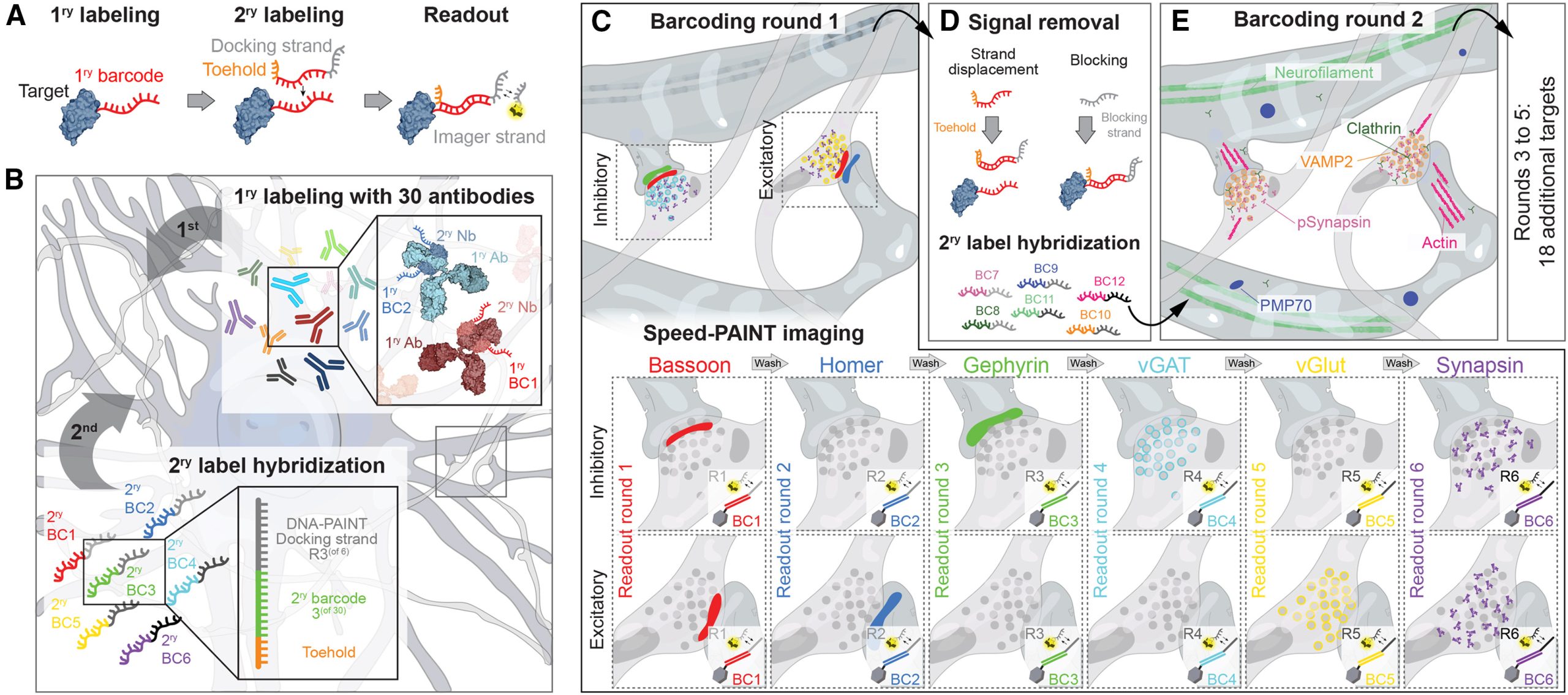
图3 基于二级标记的DNA-PAINT可实现单个蛋白的高度多重成像
结果2:
SUM-PAINT可实现亚5纳米分辨率的速度优化多重成像
研究人员使用DNA折纸术(DNA origami)评估了SUM-PAINT的性能,成功解决了5纳米间距的结构。通过对二级标记的信号消光进行有效评估,并在 DNA origami 和细胞样本中实现了超过98%的信号消光效率。这项技术还成功应用于细胞核孔复合体,展示了其在细胞标记和成像中的潜力。
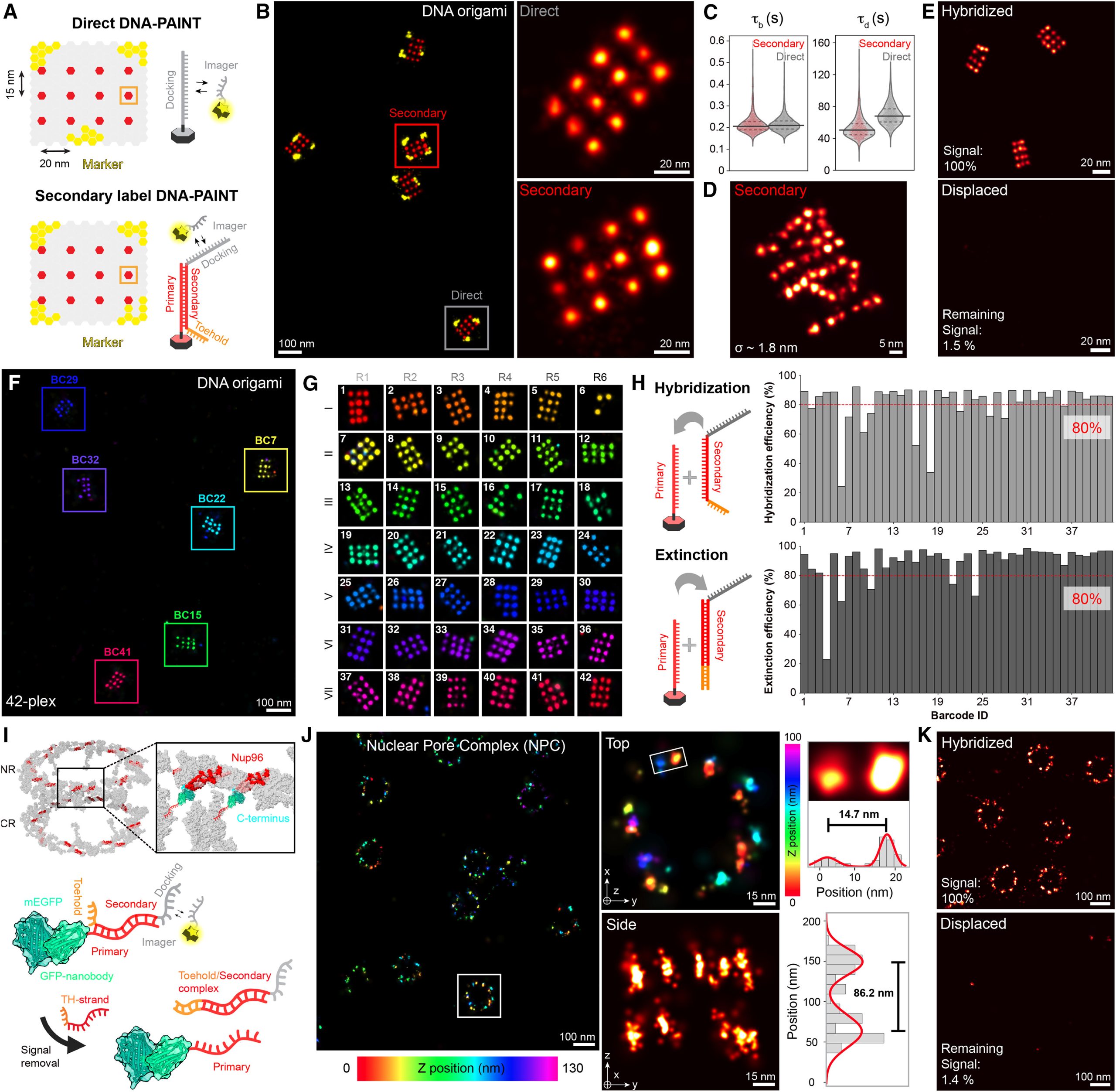
图4 SUM-PAINT可实现亚5纳米分辨率的速度优化多重成像
结果3:
单个蛋白质分辨率的30-Plex神经元图谱
研究人员使用SUM-PAINT技术构建了30种单蛋白分辨率的神经元图谱。通过对超过100种亲和试剂进行评估选择适合的抗体进行多重成像,可以在单个神经元中进行超分辨率成像,准确地展示了30种蛋白的空间分布。
该技术克服了传统方法中成像效率低下的问题,可在较短的时间内完成高效的多重成像。同时,通过综合运用信号消光方法,实现了高效的信号消光,从而为神经元细胞的高分辨率图谱构建奠定了基础。
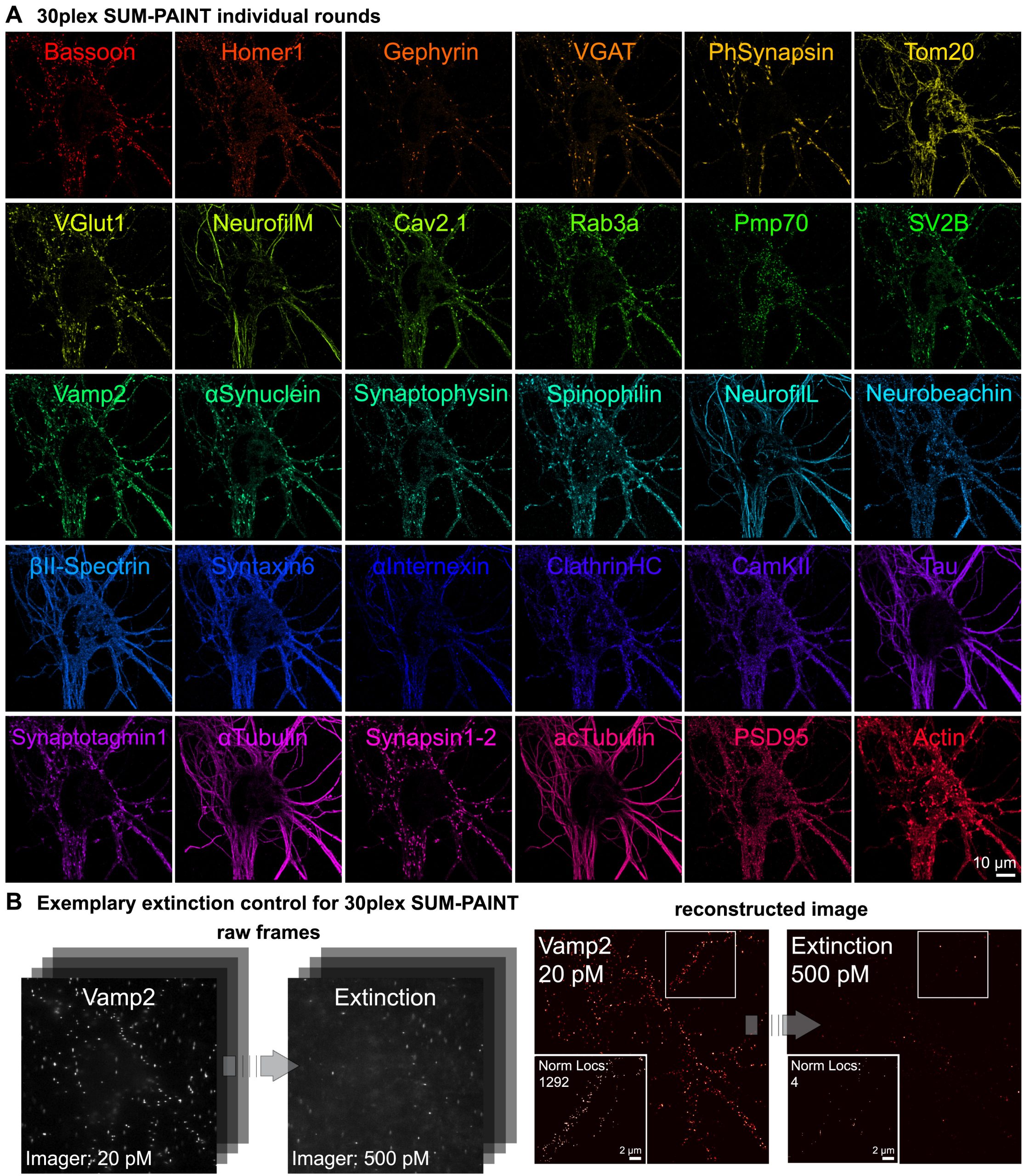
图5 30-plex SUM-PAINT实验和消光对照的各个蛋白质靶标图库
结果4:
多重SUM-PAINT技术提供纳米级的神经元结构洞察力
研究人员通过分析红细胞骨架蛋白Spectrin(βII-Spectrin)和线粒体外膜受体Tom20等结构,揭示了它们的周期性排列和三维结构。
同时,对网格蛋白(Clathrin)和神经丝蛋白等蛋白进行细致观察,研究人员发现了神经元细胞内的细微结构,如网格蛋白包被小窝(clathrin-coated pits)的大小和神经丝蛋白纤维的组织形态。
通过DNA-PAINT技术提供的单蛋白分辨率,研究人员还观察到了神经元突触中的跨突触纳米柱组织(trans-synaptic nanocolumns)和神经丝的排列方式,为神经元细胞结构提供了详细的纳米级解析。
结果5:
无监督学习应用于单蛋白分辨率的空间蛋白组学,揭示独特突触类型
研究人员利用无监督机器学习方法对单蛋白分辨率的空间蛋白质组学进行了研究,通过30个蛋白物种在六个独立数据集中的谱图,利用UMAP降维和空间聚类分析,发现了除了传统的兴奋性和抑制性突触之外的“第三种类型”,这种混合型突触具有兴奋性和抑制性标志物的混合特征。
进一步地,通过视觉检查蛋白组成,研究人员确认了该混合型突触的存在,并对其分子组成进行了详细分析,揭示了其突触小泡的再循环能力与兴奋性突触相似。
结果6:
高内涵特征空间分析可深入研究突触类型特征,揭示突触多样性
研究人员通过对多个突触蛋白进行分析(包括突触前和突触后支架蛋白、神经递质转运蛋白、突触小泡和相关蛋白等),采用UMAP进行数据降维和聚类分析,研究了这些蛋白在不同突触类型中的相对含量、空间分布和相关性,揭示了突触的多样性。
通过中心质心位置、蛋白相对量和簇体积、空间轮廓、体积相关性等指标进行分析,研究人员发现了突触类型间的差异和相似性,揭示了不同类型突触之间的蛋白组成和空间结构的特征。
同时,研究人员还讨论了特定突触类型中的蛋白簇表达水平的差异,以及突触特异性蛋白的磷酸化调节等现象。
总 结
综合来看,文章介绍了一种新颖的空间蛋白质组学技术SUM-PAINT,以及其在神经元中的应用和潜在意义,为单蛋白分辨率的全面空间蛋白质组学提供了新的思路和方法。该工作是迄今为止对突触分子多样性进行的最全面的空间研究,它结合了蛋白质特性信息和详细的形态特征。
文章讨论部分总结了SUM-PAINT的优势和潜在应用。通过将机器学习和组学方法结合到分析流程中,研究人员获得了高度多重、高分辨率的数据集,并能够深入到单个样本区域,揭示单个蛋白质的纳米排列。他们还发现了三种不同的突触亚型,其中一个是之前未报道过的,这表明了突触的纳米尺度多样性。
该研究也存在一定的局限性,例如SUM-PAINT受到亲和试剂的限制,还需要光学切片来实现对细胞的全面成像。
文章链接:
https://www.cell.com/cell/fulltext/S0092-8674(24)00248-4#%20