今年的诺奖,是AI的诺奖。
物理学奖授予了 John J. Hopfield 和AI教父 Geoffrey E. Hinton,以表彰他们利用人工神经网络进行机器学习的奠基性发现和发明。
化学奖一半授予 David Baker,以表彰他在计算蛋白质设计方面的贡献;另一半则共同授予 Demis Hassabis 和 John M. Jumper,以表彰他们在蛋白质结构预测方面的成就。
AI学者在2024年诺奖中的「大满贯」引起了广泛讨论。无论外界如何评价,不可否认的是,AI正深刻地改变着世界,推动着物理、化学、生物、医学等领域的快速发展。
今年诺贝尔化学奖的主题是「蛋白质——生命中精妙的化学工具」。诺奖官网称,今年的三位诺贝尔化学奖得主破解了蛋白质惊人结构的密码。
多年来,科学家们一直醉心于蛋白质的研究。从20世纪30年代发明的透射电子显微镜,到近10年出现的冷冻电镜,再到如今可预测蛋白质结构的人工智能系统AlphaFold2、AlphaFold3…… 随着技术进步和学科融合,研究蛋白质的方法也在不断更新和进步。
蛋白质研究如果能与AI紧密结合,势必能让我们窥见生命世界更为精彩的奥秘。
01
蛋白质的探索之路
时间拨回到20余年前。
2003年,历时13年的 “国际人类基因组计划(HGP)” 正式完成。但仅仅测绘出人类基因组序列并非这一计划的最终目的,必须对作为其编码产物的蛋白质组进行系统深入的研究,才能真正实现基因诊断和基因治疗。
因此,在HGP完成之际,国际人类蛋白质组计划(HPP)随之启动,首批行动分 “人类肝脏蛋白组计划” 和 “人类血浆蛋白质组计划” 两部分同步进行。
2010年,人类蛋白质组组织(HUPO)再次正式启动HPP,旨在为全球合作、数据共享、质量保证和增强基因组编码蛋白质组的准确注释创建一个框架。正式启动后的这10余年里,该计划取得了诸多成就。
2014年,研究人员使用高分辨率傅里叶变换质谱法提出了人类蛋白质组的绘制图,对30个组织学正常的人类样本进行了深入的蛋白质组学分析,鉴定出由17000多个基因编码的蛋白质,约占人类注释蛋白质编码基因总数的84%。这是人类蛋白质组的第一张草图。
随后,2020年(即HPP正式启动后十周年之际),这项计划取得了里程碑式的进展:精确地覆盖了90.4%的人类蛋白质,这一成果为疾病预防和个体化医学提供了重要思路。
此后,HPP继续致力于鉴定剩下10%的人类蛋白质。但是后续的研究或许会更加困难和缓慢,因为许多特殊蛋白质要么只在特定和难以获得的组织类型中表达,要么很难使用传统的蛋白质组学技术对其进行分离和分析。
即使90%的蛋白质已被鉴定,许多蛋白质的生物学功能仍未得到充分研究。正如HPP主席 Robert Morit 指出的,大多数研究集中在少数容易检测的蛋白质上,而大量蛋白质仍处于 “未解密” 状态。
我们需要一个新的计划,来研究这些鲜有人涉及的未充分研究的蛋白质——
2022年,六国科学家联合发起了一项名为 “未充分研究的蛋白质计划(Understudied Proteins Initiative)” 的调查。他们呼吁科学家们解密那些未知的 “暗物质” 蛋白质,以不断扩大人类对生物大分子认知的边界。
02
「未被充分研究的蛋白质」
在蛋白质科学家看来,现有的蛋白质组学研究中,95%的论文关注的是5000种已充分研究的人类蛋白质,而大量与疾病相关的未表征蛋白质仍然缺乏深入研究。
据美国食品与药品监督管理局(FDA)批准药物的作用靶点的分析,“目前只有5-10%的潜在靶标蛋白质被加以开发”。
为了解决这些重要蛋白质的相关研究 “坐冷板凳” 的问题,“未充分研究的蛋白质” 计划应运而生。Nature Methods 和 Nature Biotechnology 上同时发表了两篇重要文章,呼吁学界通过系统地关联未表征的蛋白质和已知功能的蛋白质,缩小两者之间的注释差距,从而为详细的机制研究奠定基础。


“未充分研究的蛋白质计划” 发起者及其科研单位:
Georg Kustatscher: University of Edinburgh, Edinburgh, UK
Tom Collins: Wellcome Trust, London, UK
Anne-Claude Gingras: University of Toronto, Toronto, Ontario, Canada
Tiannan Guo: Westlake University, Hangzhou, China
Henning Hermjakob: EMBL-EBI, Cambridge, UK
Trey Ideker: University of California San Diego, La Jolla, CA, USA
Kathryn S. Lilley: University of Cambridge, Cambridge, UK
Emma Lundberg: KTH-Royal Institute of Technology, Stockholm, Sweden
Edward M. Marcotte: University of Texas at Austin, Austin, TX, USA
Markus Ralser: Charité University Medicine, Berlin, Germany;
Juri Rappsilber: Technische Universität Berlin, Berlin, German
未被充分研究的蛋白质到底有多重要?计划的发起者认为,对它们进行研究,一方面有助于我们对基本的生命规律理解;另一方面,从药物发现的角度来看,能提高我们对蛋白质与小分子以及蛋白与蛋白间相互作用的理解,从而更好地指导药物开发工作。
然而,传统研究手段在这些领域的探索方法和效率有限。令人欣慰的是,随着技术的进步,新的工具正在悄然改变这一格局。
03
AI赋能的蛋白质组学研究
2020年,DeepMind开发的AlphaFold2成功破解了蛋白质结构预测这一困扰人类半个世纪的难题,引起了全球生命科学领域的强烈反响。到2022年,AlphaFold2已预测出超过100万个物种的2.14亿个蛋白质结构,几乎涵盖了DNA数据库中已知生物的所有蛋白质。
今年5月,AlphaFold3也随之问世(Demis Hassabis 和 John M. Jumper两位新晋诺奖得主为主要研究者)。这一AI模型能够准确预测蛋白质、DNA、RNA以及配体等所有生命分子的结构及其相互作用方式,是继AlphaFold2之后的又一重大突破。
至此,蛋白质的结构预测取得了巨大突破。AI不仅改变了蛋白质结构的预测方式,还为进一步探索这些蛋白质的功能提供了强大的工具。例如,通过赋能蛋白质组学,推动功能研究的前沿进展。
AI的进步,尤其是机器学习和深度学习技术的应用,正在为蛋白质组学研究带来革命性的变化,特别是在数据分析、蛋白质鉴定和生物标志物发现等方面。
例如,通过机器学习和深度学习技术,质谱数据处理变得更加高效,肽段和蛋白质鉴定的准确性也显著提升。此外,AI优化了蛋白质组学的工作流程,从肽段识别到定量分析,每个环节都变得更加精准和高效,为精准医学和临床应用提供了强有力的支持。
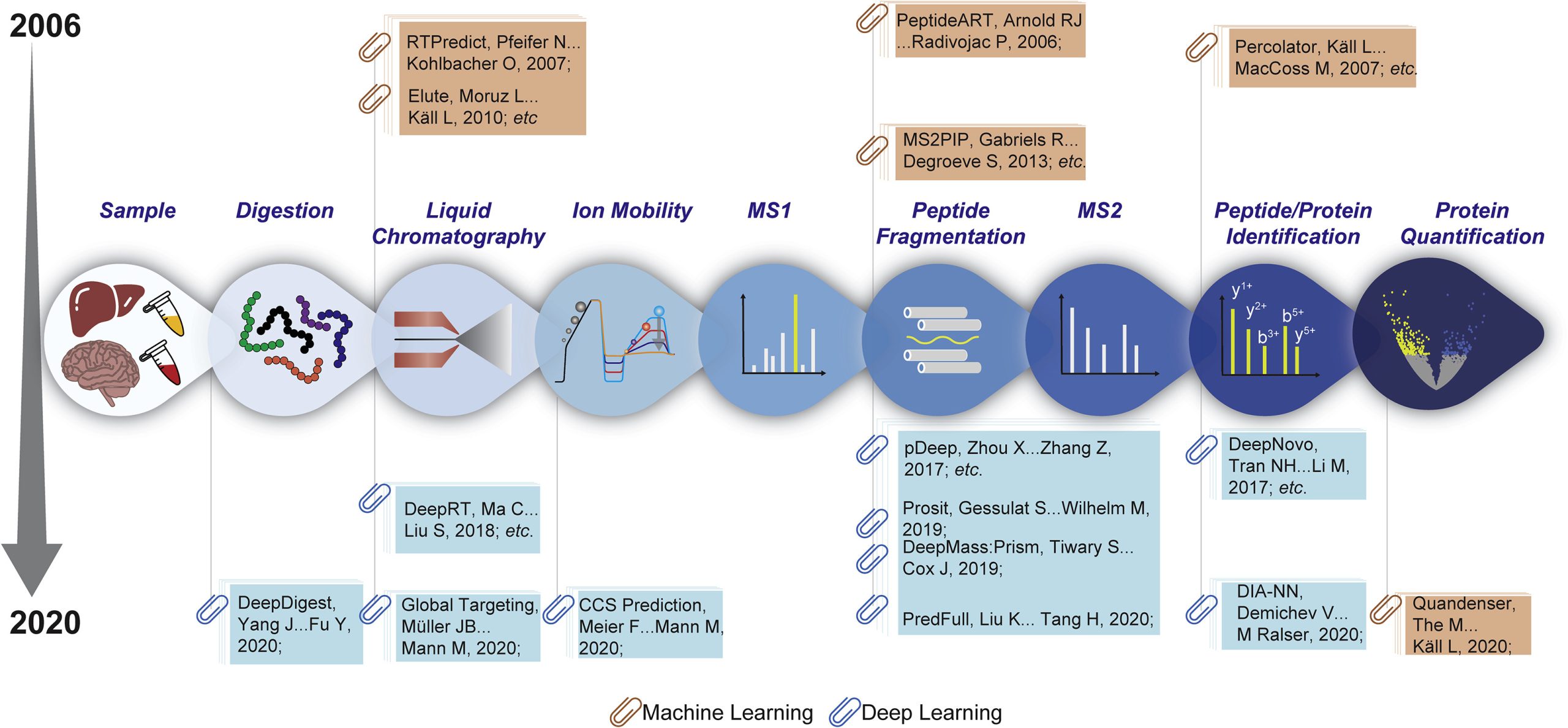
AI几乎应用于蛋白质组学工作流程的每个步骤
同时,AI还能挖掘数据中隐藏的复杂模式,帮助发现新的生物标志物,为疾病诊断和治疗提供新的方向。
2022年,西湖大学生命科学学院郭天南团队、工学院李子青团队,联合多个国内外临床单位,通过AI算法结合蛋白质组特征,构建了一个可以用于甲状腺结节良恶性评估的深度学习模型。在发现集中,该模型对甲状腺结节良恶性的评估准确率高达91%,在回顾性和前瞻性测试集中的评估准确率也分别高达89%和85%。该研究展示了高通量蛋白质组学与AI技术的深度碰撞。

AI还促进了蛋白质组学与其他组学领域(如基因组学、代谢组学)以及生物医学数据(如临床记录和成像数据)的整合。例如,在癌症研究中,AI可以将基因突变数据与蛋白质组学数据结合,帮助识别那些可能被突变影响的关键蛋白质。
这种数据整合能力使得AI成为了多组学研究中的重要工具,也加速了研究人员对生物系统全局性的理解。
▼
AI技术不仅改变了蛋白质结构预测的方式,也极大地推动了蛋白质功能的研究和应用,在对 “未充分研究蛋白质” 的探索中展现出了巨大潜力。
随着AI在蛋白质组学中的深度融合,我们有理由相信,未来的生命科学研究将更加精准和高效,为疾病的诊断、治疗和新药开发带来新的希望。
AI不因诺奖而「热」,但生命科学研究中的AI正在发光发热。如何穿越层层迷雾,找到人体蛋白质背后隐藏的生命奥秘?且看科学家如何善假于物,拨云见日。
参考资料:
1. A high-stringency blueprint of the human proteome.Nature.
https://www.nature.com/articles/s41467-020-19045-9#Fig3
2. A draft map of the human proteome.NIH.
https://pubmed.ncbi.nlm.nih.gov/24870542/
3. https://understudiedproteins.org/survey
4. https://understudiedproteins.org/conference
5. Understudied proteins: opportunities and challenges for functional proteomics. Nature Methods.
6. Mass spectrometry-based protein identification by integrating de novo sequencing with database searching - PMC (nih.gov)
7. Artificial intelligence for proteomics and biomarker discovery. Mann, Matthias et al. Cell Systems, Volume 12, Issue 8, 759 - 770
8. Artificial intelligence defines protein-based classification of thyroid nodules. Sun, Y., Selvarajan, S., Zang, Z. et al. Cell Discov 8, 85 (2022).