分子靶向药物(尤其是激酶抑制剂(Kinase inhibitors))已经在多种癌症的临床治疗中取得了成功,但在某些癌症类型中(如肉瘤)分子机制仍不够清楚。肉瘤是一种罕见的癌症,其致癌驱动因子通常不清楚。药物的分子机制受到细胞环境的影响,因此在特定细胞系统中了解药物的MoA对成功使用它作为药物至关重要。
先前的研究主要集中在记录多个癌症细胞系的基因组和转录组,并通过与药物敏感性数据相关联来解读这些信息。最近,这些研究已扩展到蛋白质组的水平,为癌症研究提供了重要的公开资源。

图1 论文截图
2023年12月18日,德国慕尼黑工业大学的 Bernhard Kuster 团队在 Molecular Systems Biology 发表了新的文章 Illuminating phenotypic drug responses of sarcoma cells to kinase inhibitors by phosphoproteomics。
文章通过对17种肉瘤细胞系的表型、蛋白质组和磷酸化蛋白组进行全面分析,揭示了细胞系间药物反应的差异、细胞内蛋白质和磷酸化模式的多样性,并鉴定了与药物敏感性相关的关键生物标志物,为癌症个性化治疗提供了深入的分子基础。
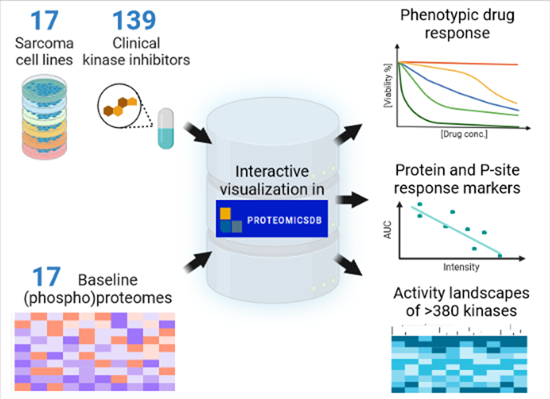
图2 图文概要
研究样本及方法
通过对17个肉瘤细胞系的蛋白质组和磷酸蛋白组的分析,以及对150种癌症药物的筛选,揭示肉瘤细胞对分子靶向药物的表型反应。
研究结果
1. 三个系统性数据集
研究生成了三个系统性数据集,包括:药物剂量-响应数据集:对17个细胞系进行了150种癌症药物(包含139种激酶抑制剂)的剂量依赖性筛选,生成了2550个药物剂量-响应曲线。基线蛋白组数据集:记录了17个细胞系的基线(即未经药物处理的)全蛋白组数据,每个细胞系量化了9200-10,200个蛋白质组。基线磷酸化蛋白组数据集:记录了17个细胞系的基线磷酸化蛋白质组数据,量化了每个细胞系的10,200-27,200个磷酸化位点。
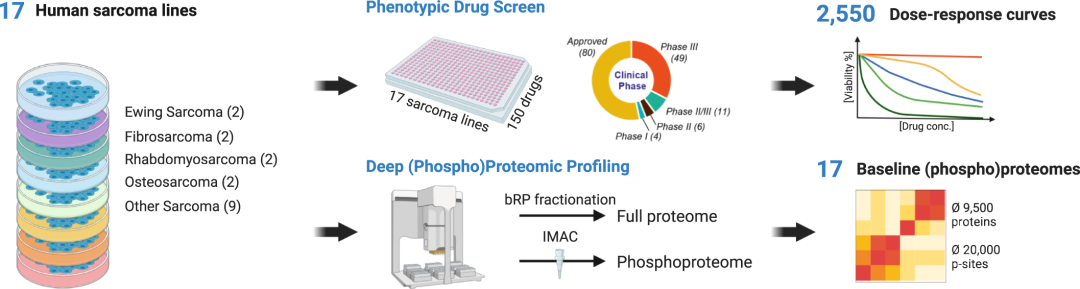
图3 实验和数据分析工作流程的示意图
2. 肉瘤细胞系对激酶抑制剂表现出不同的反应
通过对药物剂量-响应数据进行无监督聚类,结果显示很多激酶抑制剂(KIs)对肉瘤细胞并没有实质性地抑制肉瘤系的生长,这可能因为其中许多Kis靶向的特定激酶和信号通路并不是这些细胞的致癌驱动因子。相比之下,针对细胞生存的通用机制的药物(如DNA复制、转录和细胞周期抑制剂)通常强烈抑制大多数细胞系的增殖。比如孤儿药Volasertib(一种干扰有丝分裂的PLK1抑制剂) 对许多肉瘤细胞系都有效,可能为肉瘤的额外临床试验提供了合理的依据。PI3K抑制剂Copanlisib也显示出对大多数肉瘤细胞系的高效力,但筛选中的大多数其他PI3K抑制剂没有显示出实质性的作用,这意味着Copanlisib的未知脱靶可能是其对肉瘤细胞抑制作用的原因。
研究人员对其他药物簇也进行了详细的分析表明,表型筛选对于识别具有重新利用潜力的高级临床化合物是强有力的,但某些细胞系对这些药物有或没有反应的原因往往仍然难以捉摸。
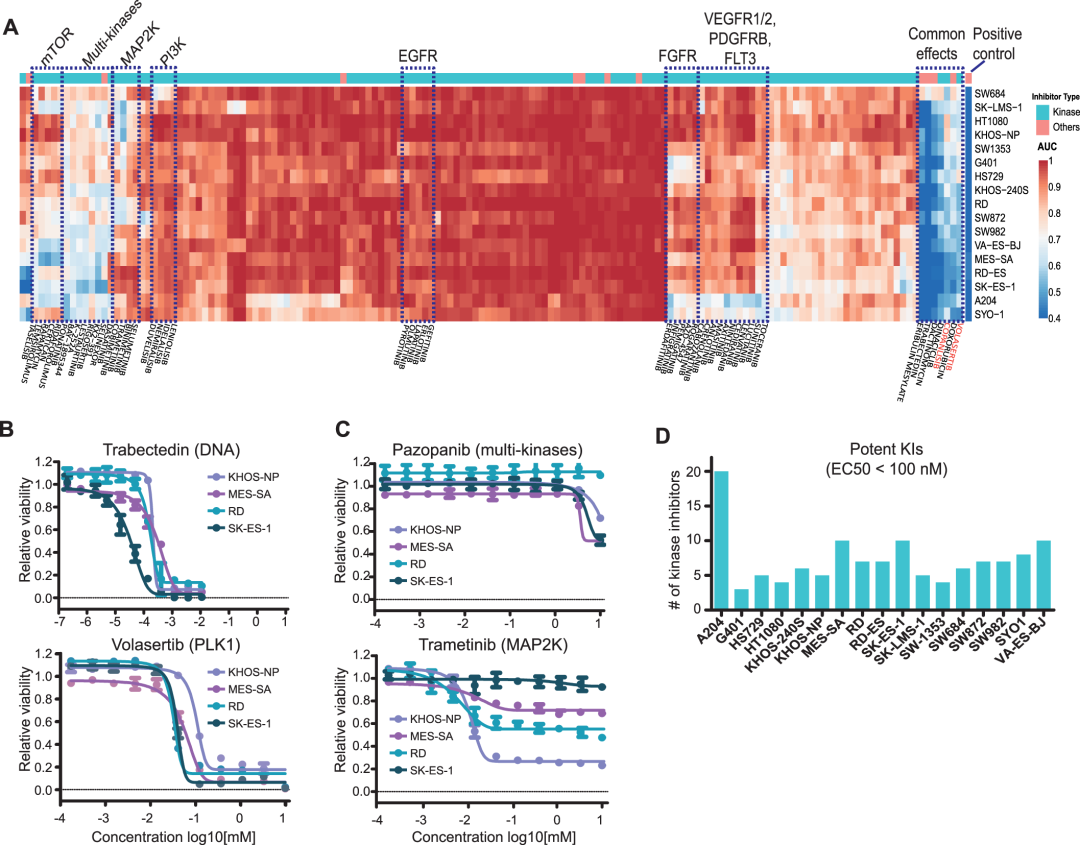
图4 表型药物筛选揭示了肉瘤细胞系常见且独特的药物反应。热图(A)总结了 17 种肉瘤细胞系(行)对 150 种临床癌症药物(列)的反应
3. 分子活动景观解释药物反应并提示有效药物
为了在分子水平上更好地理解上述药物反应,将药物的表型剂量-响应特性与基线(即未经治疗的细胞系)的蛋白质组和磷酸化蛋白质组整合,研究建立了383种激酶在每个细胞系中的活性评分。这一评分通过综合激酶丰度和磷酸化等信息,形成了细胞系的活性景观。对这些活性分数进行排序为每个细胞系提供了哪些药物可能能够抑制其增殖的假设。
实验结果表明,在许多情况下,这些假设能够得到验证。通过此分析,研究还指出了蛋白质组和磷酸化蛋白质组数据在解释药物敏感性方面的重要性,同时提出了一种选择对特定细胞系有效药物的合理策略。需要注意的是,蛋白质组和磷酸化蛋白质组数据中计算的高或低激酶活性评分不一定与药物屏幕中测得的表型(如细胞存活)相关,因为这些激酶可能在细胞中担任其他重要功能。
4. 通过蛋白质组表达谱识别在细胞周期特定步骤起作用的药物
研究人员基于先前研究报道的在细胞周期间差异表达的119个蛋白质估计了细胞在特定细胞周期阶段的比例,并对这119个“周期蛋白质(periodic proteins)”的表达与87种药物的反应(最小AUC <0.8)进行了相关分析,发现细胞周期预测比例与对应药物报道的一致。例如,CDK抑制剂Dinaciclib和DNA复制抑制剂Doxorubicin在S期表现出负相关,而PI3K/mTOR抑制剂在M期呈现正相关,表明M期细胞对这些抑制剂具有耐药性。
5. 具有相关作用机制(MoA)的药物共享反应标志物
研究人员通过系统分析,使用SMBPLSR算法发现了12个药物簇,这些药物在细胞系中显示出相似的反应,并且它们的反应与多个蛋白质和磷酸化位点的丰度相关。其中三个在下图中做了说明。
第一个簇包含四个MAP2K抑制剂和药物敏感性标志,包括已知是MAPK途径成员的蛋白质和磷酸化位点,从而验证了这一方法。
第二簇包含PI3K和mTOR抑制剂,标志物包括RRM2(核糖核苷酸还原酶家族成员2) 的T33磷酸化和Lamin A的T394磷酸化,表明对于这些药物,细胞周期中的G2/M期与其敏感性和耐药性相关。
第三簇包含AURK和PARP抑制剂,发现BCL11B的S678磷酸化与敏感性相关,CD44的S706磷酸化和蛋白水平与耐药性相关。这些标志物的发现有望帮助理解药物的作用机制和预测细胞对药物的响应。
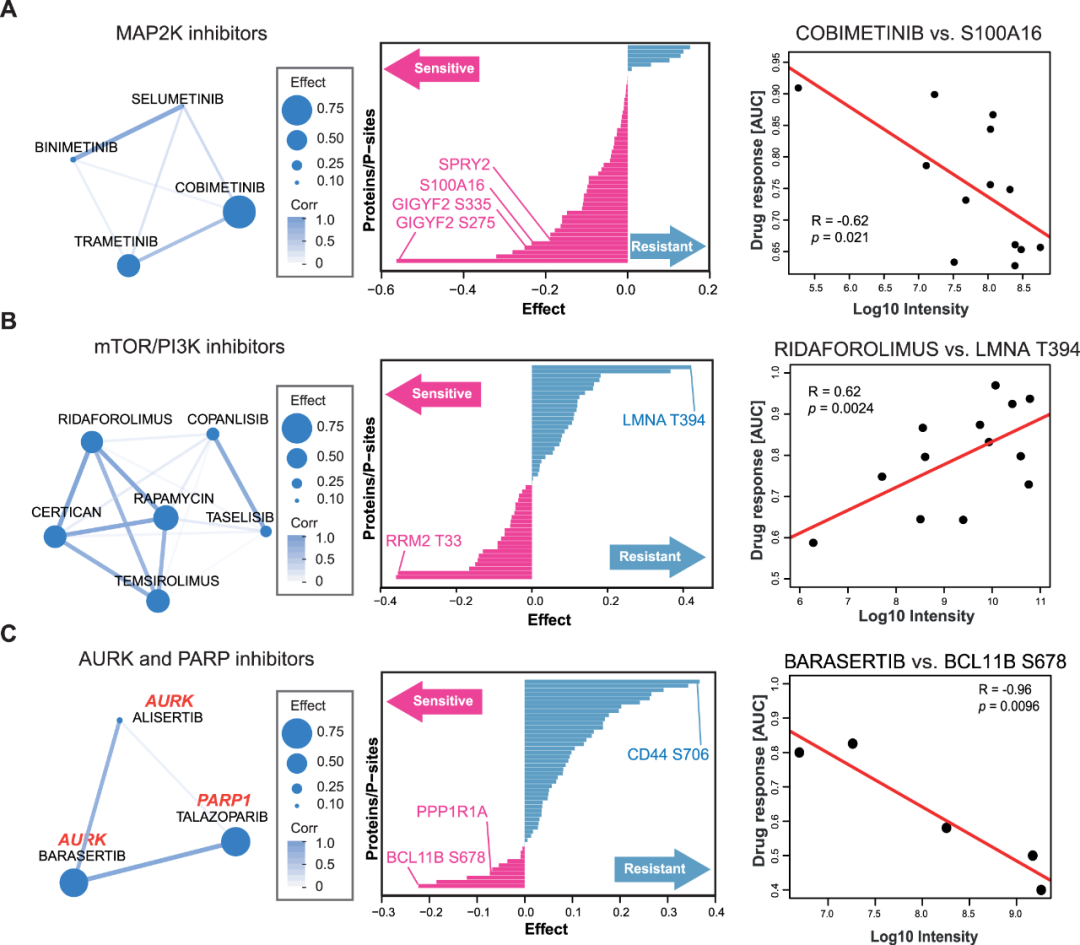
图5 具有相关 MoA 的药物具有共同的药物反应标志物
6. 相关表型和蛋白型鉴定药物反应的标志物
通过相关表型和蛋白型,研究人员确定了与药物反应相关的标志物。他们应用弹性网络回归模型(Elastic net)对 150 种药物、12,000 种蛋白质和 53,000 个磷酸位点产生的 970 万个关联进行了分析。
以Cobimetinib和Infigratinib为例,研究人员成功识别了与药物敏感性和耐药性相关的蛋白质和磷酸化位点。例如,MAP2K抑制剂Cobimetinib在MAPK信号通路中表现出多个敏感性标志物,而FGFR抑制剂Infigratinib的敏感性标志物包括FGFR信号通路中关键蛋白FRS2的磷酸化位点。这些结果揭示了药物反应的潜在分子机制。
7. Infigratinib通过抑制FGFR下调ERBB2和TOP2B的磷酸化
在使用相关和回归方法进行分析时,虽然能够获得有力的结果,但并不一定意味有因果关系。为了跟进前一部分的一些观察结果,研究人员在FGFR2过表达和FGFR2抑制剂敏感的腭部肉瘤细胞系G401中,以时间依赖的方式测量了其在Infigratinib处理过程中的磷酸化蛋白质组。
通过分析这一过程,研究人员发现Infigratinib抑制作用下,ERBB2和TOP2B的磷酸化水平下调。这一实验证实了之前由Elastic net回归提出的假设。结果还表明,TOP2B在Mitosis过程中发挥着重要作用,与多种药物的效应在Mitosis水平上汇聚。最后,研究人员观察到ERBB2/HER2和FGFR信号通路之间存在功能性联系,这一发现对解释由Elastic net回归提出的假设和后续实验证明提供了理论基础。
论文最后指出,这项研究为实现个性化治疗奠定了基础,但仍需更深入的研究来理解不同细胞系的分子特征和药物反应之间的关系,未来研究方向包括更多数据集的整合、系统性生物信息学分析、临床验证等。
文章链接:
https://www.embopress.org/doi/full/10.1038/s44320-023-00004-7









