
2023年11月15日,德国马克斯普朗克生化研究所的 Matthias Mann 团队在 Nature 发表了新的文章:The social and structural architecture of the yeast protein interactome。
文章通过整合深度学习结构预测技术,详细解析了酵母蛋白相互作用网络的社交结构,并揭示了多个未知蛋白质复合物的结构,为理解蛋白质相互作用及其在细胞生物学中的作用提供了新的见解。
本文介绍了一种用于映射酿酒酵母(Saccharomyces cerevisiae)蛋白相互作用组的高灵敏度高通量方法。研究者使用亲和纯化质谱(AP-MS)方法,结合定量的二维分析策略,全面地绘制了酿酒酵母的相互作用组。
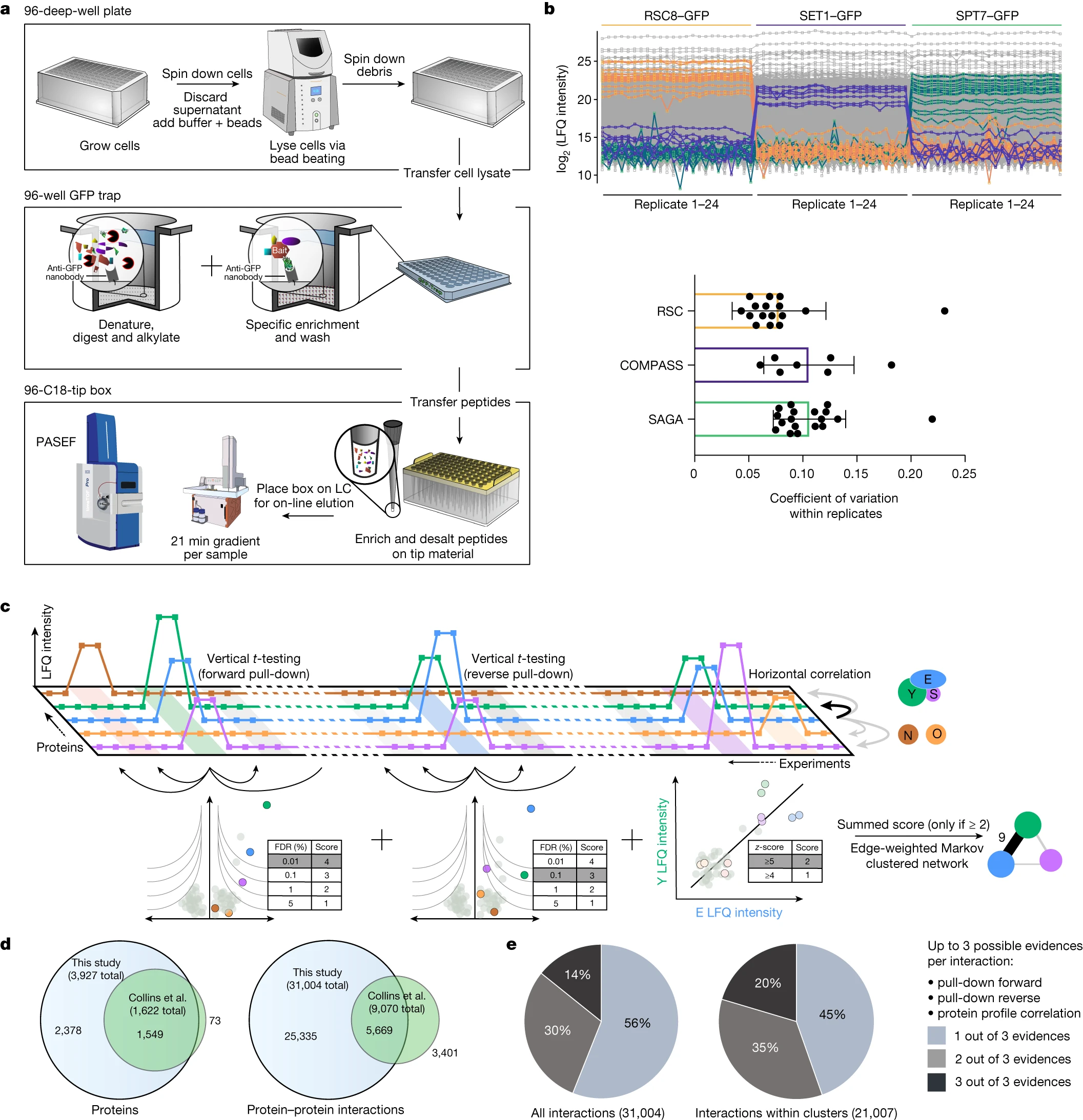
图1 一种全面且可扩展的相互作用组学技术
研究人员回顾了过去20多年来使用基于质谱的蛋白质组学对细胞相互作组进行大规模研究的历史。其中有两项研究成功地纯化了近一半的表达酵母蛋白质组,并确定了相互作用因子。这些数据集已被广泛挖掘,形成了基于网络的细胞蛋白质组视图。
基于相互作用组对于功能理解的重要性以及过去十年间质谱技术的重大改进,研究人员生成了一个在给定状态下包含所有蛋白质的相互作用组。他们使用了内源GFP标记的酵母库,其中包含在标准生长条件下可检测的4,159个蛋白质。通过对工作流程的小型化和标准化,结合一个高度稳定的液相色谱系统和高灵敏度的捕集离子迁移谱(trapped ion mobility spectrometry,TIMS),研究者取得了非常高的数据完整性。该工作流程仅需1.5毫升酵母培养液,每天可保持恒定的60个pull-down,可用于具有不同丰度的可溶或膜蛋白的相同条件(图1a)。
通过对96孔板中的内源GFP标记的蛋白质库的复制分析,研究者成功生成了包含3,927个蛋白质和31,004个相互作用的高度结构化网络。这个互作组网络涵盖了整个表达的酵母蛋白质组,其中包括数量很少的表观遗传复合物、器官膜复合物和通过丰度相关性推断的不可标记复合物。
通过这一 “饱和” 的相互作用组,研究者发现绝大多数酵母蛋白质高度相互连接,平均每个蛋白质有16个相互作用者,中位数为6,明显多于人类 BioPlex 相互作图谱(平均相互作用8)。与人类社交网络相似,蛋白质之间的平均最短距离为4.2次相互作用(interactions)。AlphaFold-Multimer 为先前未经表征的蛋白质提供了新的功能洞察。
为了便于大家对感兴趣的相互作用进行探索,研究者还创建了一个Web门户(www.yeast-interactome.org),该门户提供了有关蛋白质-蛋白质关联的统计证据,并总结了所得到的簇。
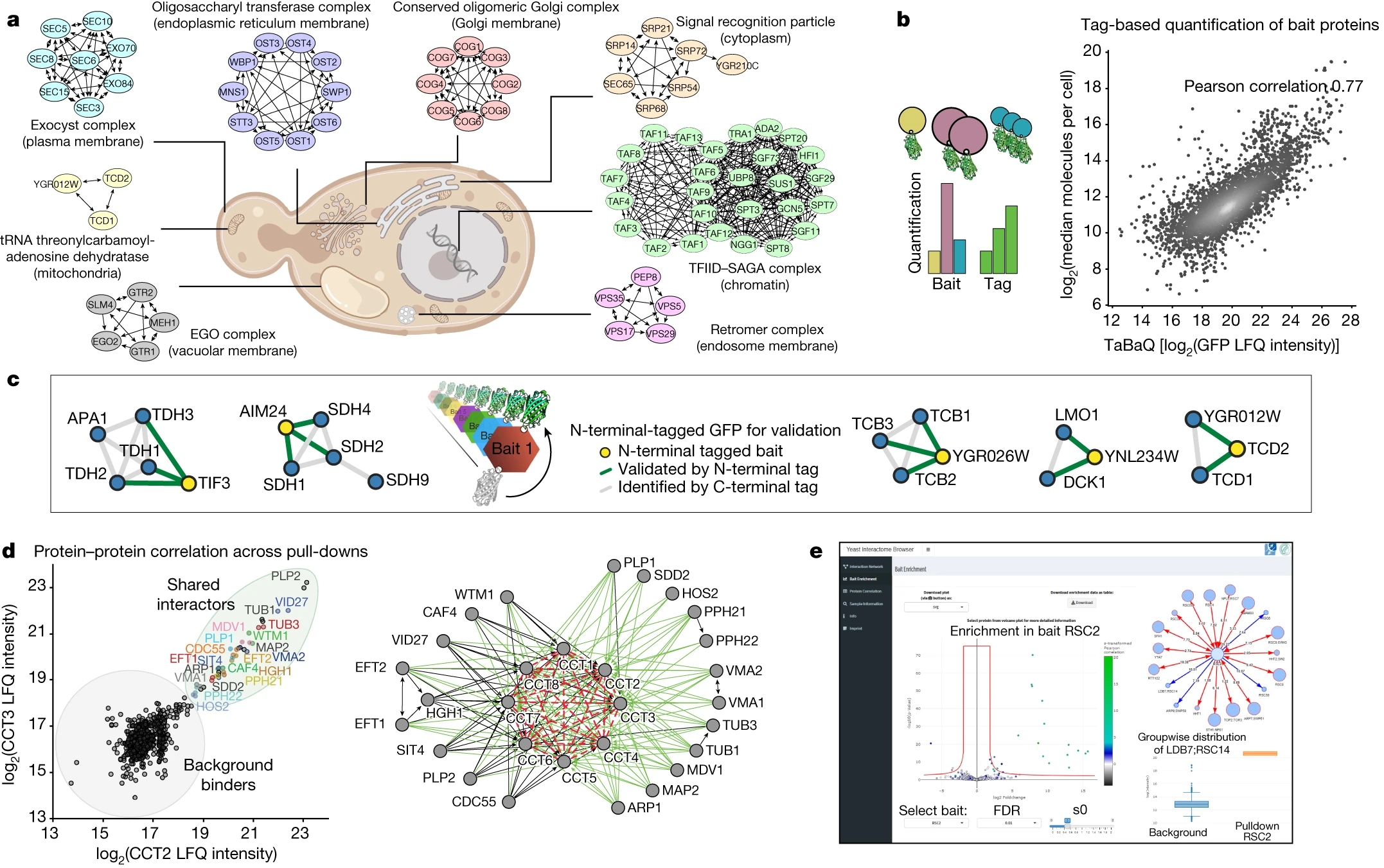
图2 用于探索相互作用组的高质量数据集
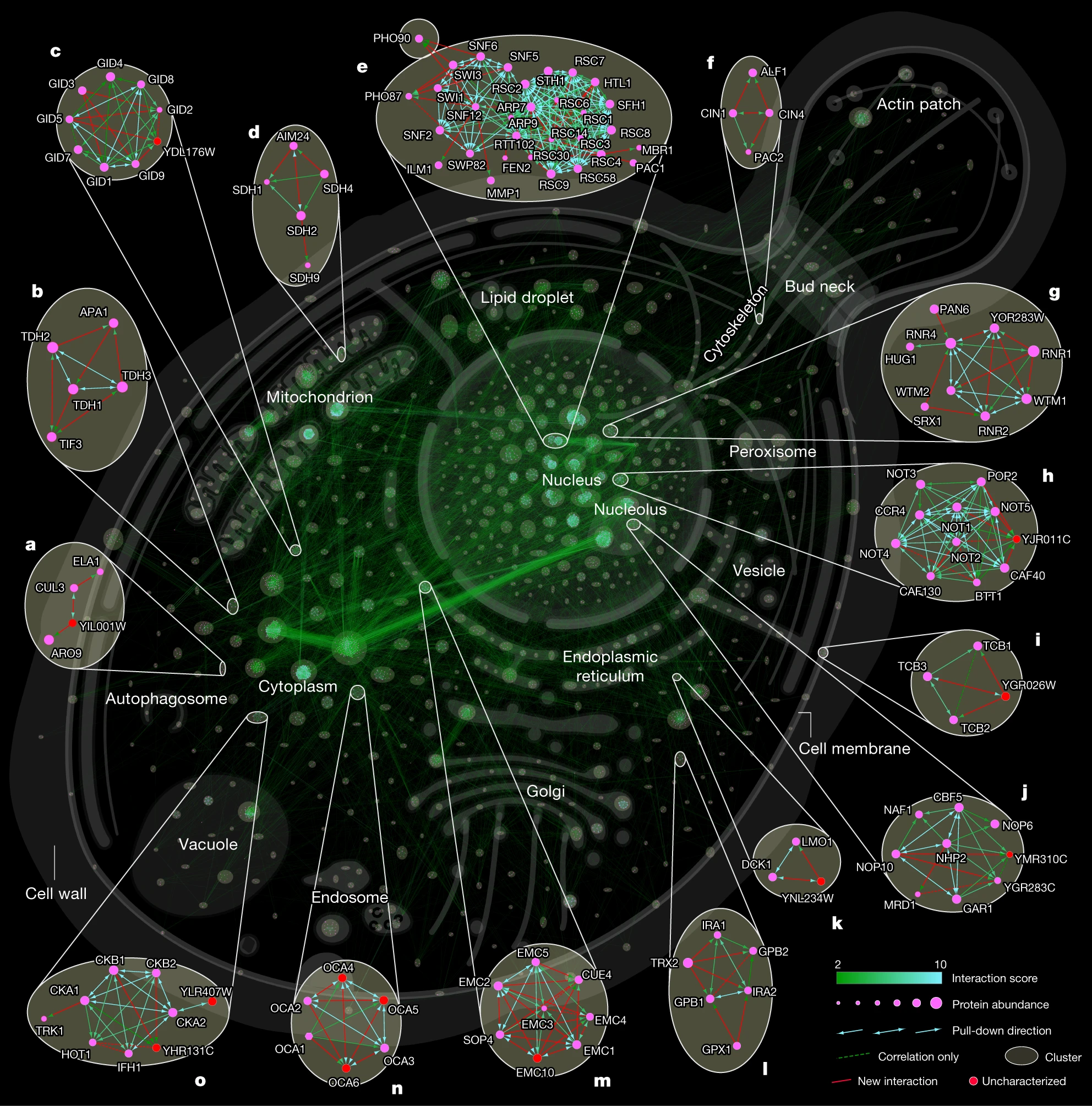
图3 所有重要相互作用的细胞间相互作用图
本研究的相互作图谱中包含了未经表征的蛋白质或 BioGRID 数据库中没有报道的相互作用,从而提供了新的生物学见解。例如,文章确定了未知功能蛋白Cue4与内质网膜复合物(EMC)之间的相互作用;发现了两个未经表征的蛋白YPR063Cp和YNR021Wp之间的新的相互作用,等等。
研究者还发现了一个新的复合物,由三个未报告的相互作用定义(所有的相互作用得分都是最大的10),这些相互作用连接了Tcd1、Tcd2(参与tRNA碱基修饰的线粒体蛋白)和YGR012Wp(一种未知功能的蛋白,作者将其命名为Tcd3)。Tcd1和Tcd2的同源物在大肠杆菌(TcdA)中功能于三蛋白质复合物中,参与了所有三个生命域,包括人类。
总的来说,研究者开发并应用了一种新颖且高度可扩展的相互作用组技术,能够以之前所需的测量时间和起始材料的一小部分来重复测量酵母网络。他们的研究几乎达到了饱和,并包含了在实验条件下预期的几乎所有复合物。
研究表明,高置信度的相互作用数据为最近开发的深度学习模型提供了理想的基础,这些模型可以从序列中预测复合物结构,产生与功能相关的全新结构模型。由此得到的网络的高连接性在酵母蛋白质之间的平均最短路径为4.2,范围从高度连接的蛋白质只需三个步骤到连接较少的蛋白质,平均需要超过7个步骤。
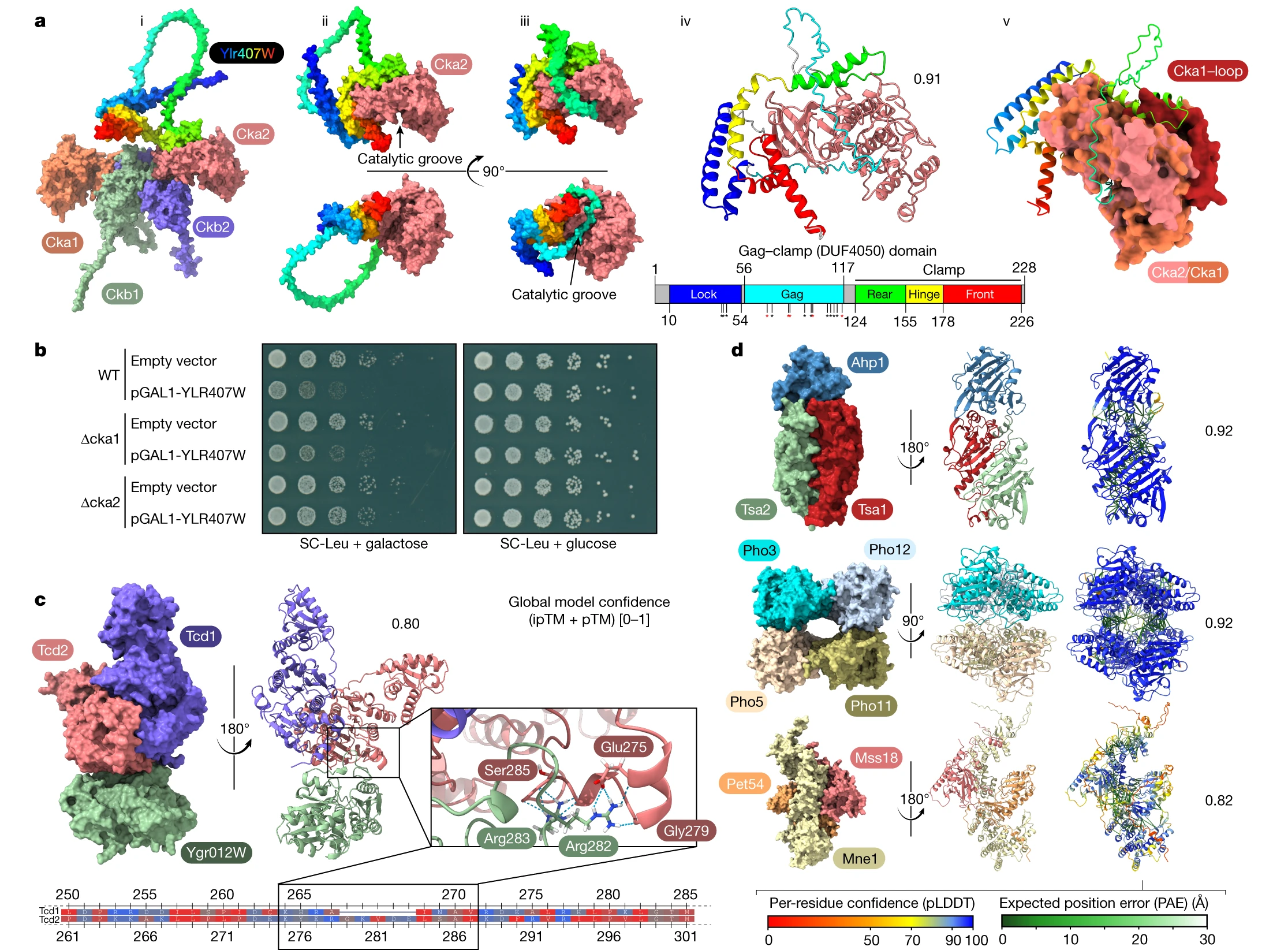
图4 基于相互作用的结构预测
研究者展望这种工作流程可以轻松地用于其他内源标记的模型生物体或用于研究动态生物过程或扰动存在下的相互作组的重塑。他们还设想将这种相互作组数据与其他相互作用技术结合使用,比如BioID或APEX,使用可以通过SWAp-Tag等平台轻松生成的标记库。全面的酵母相互作组数据可以进一步用作蛋白质复合物的假设驱动分析的先验知识,例如用于本地蛋白质复合物的共分馏质谱(Co-Fractionation coupled to mass spectrometry, coFrac-MS)分析。此外,研究者想象这样的相互作组数据也可以与质谱交联研究相结合。
通过整合深度学习的结构预测技术,研究者不仅揭示了酵母蛋白相互作用网络的社交结构,还展示了一些未知蛋白质复合物的结构。这些新的结构预测为未知蛋白质的功能提供了独特的见解,并且为未来的生物学实验和功能研究提供了有价值的线索。这项工作对于深入了解蛋白质相互作用及其结构在细胞生物学中的作用具有重要意义。
论文链接:
https://www.nature.com/articles/s41586-023-06739-5
相关研究延伸阅读
2015年,Mann 等人在 Cell 发文 A Human Interactome in Three Quantitative Dimensions Organized by Stoichiometries and Abundances。他们使用一种新的质谱定量方法,确定了每一种蛋白相互作用的强度,构成了构成了详细而复杂的人类蛋白“社交网络”:5400多个蛋白质,28000种蛋白间的相互作用。

论文链接:https://www.cell.com/cell/fulltext/S0092-8674(15)01270-2?_returnURL=https%3A%2F%2Flinkinghub.elsevier.com%2Fretrieve%2Fpii%2FS0092867415012702%3Fshowall%3Dtrue
2017年,美国哈佛医学院的 J. Wade Harper 等人在 Nature 发文 Architecture of the human interactome defines protein communities and disease networks。他们绘制出人类5800多个基因编码的蛋白相互作用图谱,是当时最大的人类蛋白质相互作用图谱。

论文链接:https://www.nature.com/articles/nature22366









